+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8jr0 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in complex with TBAJ-587 | |||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / ATP synthase / Mycobacterium tuberculosis / cryo-EM | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton motive force-driven plasma membrane ATP synthesis / proton motive force-driven ATP synthesis / proton-transporting two-sector ATPase complex, proton-transporting domain / proton-transporting ATPase activity, rotational mechanism / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / peptidoglycan-based cell wall / ADP binding / hydrolase activity ...proton motive force-driven plasma membrane ATP synthesis / proton motive force-driven ATP synthesis / proton-transporting two-sector ATPase complex, proton-transporting domain / proton-transporting ATPase activity, rotational mechanism / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / peptidoglycan-based cell wall / ADP binding / hydrolase activity / lipid binding / ATP hydrolysis activity / extracellular region / ATP binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  | |||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Zhang, Y. / Lai, Y. / Liu, F. / Rao, Z. / Gong, H. | |||||||||||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Inhibition of M. tuberculosis and human ATP synthase by BDQ and TBAJ-587. 著者: Yuying Zhang / Yuezheng Lai / Shan Zhou / Ting Ran / Yue Zhang / Ziqing Zhao / Ziyan Feng / Long Yu / Jinxu Xu / Kun Shi / Jianyun Wang / Yu Pang / Liang Li / Hongming Chen / Luke W Guddat / ...著者: Yuying Zhang / Yuezheng Lai / Shan Zhou / Ting Ran / Yue Zhang / Ziqing Zhao / Ziyan Feng / Long Yu / Jinxu Xu / Kun Shi / Jianyun Wang / Yu Pang / Liang Li / Hongming Chen / Luke W Guddat / Yan Gao / Fengjiang Liu / Zihe Rao / Hongri Gong /   要旨: Bedaquiline (BDQ), a first-in-class diarylquinoline anti-tuberculosis drug, and its analogue, TBAJ-587, prevent the growth and proliferation of Mycobacterium tuberculosis by inhibiting ATP synthase. ...Bedaquiline (BDQ), a first-in-class diarylquinoline anti-tuberculosis drug, and its analogue, TBAJ-587, prevent the growth and proliferation of Mycobacterium tuberculosis by inhibiting ATP synthase. However, BDQ also inhibits human ATP synthase. At present, how these compounds interact with either M. tuberculosis ATP synthase or human ATP synthase is unclear. Here we present cryogenic electron microscopy structures of M. tuberculosis ATP synthase with and without BDQ and TBAJ-587 bound, and human ATP synthase bound to BDQ. The two inhibitors interact with subunit a and the c-ring at the leading site, c-only sites and lagging site in M. tuberculosis ATP synthase, showing that BDQ and TBAJ-587 have similar modes of action. The quinolinyl and dimethylamino units of the compounds make extensive contacts with the protein. The structure of human ATP synthase in complex with BDQ reveals that the BDQ-binding site is similar to that observed for the leading site in M. tuberculosis ATP synthase, and that the quinolinyl unit also interacts extensively with the human enzyme. This study will improve researchers' understanding of the similarities and differences between human ATP synthase and M. tuberculosis ATP synthase in terms of the mode of BDQ binding, and will allow the rational design of novel diarylquinolines as anti-tuberculosis drugs. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8jr0.cif.gz 8jr0.cif.gz | 811.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8jr0.ent.gz pdb8jr0.ent.gz | 672.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8jr0.json.gz 8jr0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8jr0_validation.pdf.gz 8jr0_validation.pdf.gz | 1.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8jr0_full_validation.pdf.gz 8jr0_full_validation.pdf.gz | 1.8 MB | 表示 | |
| XML形式データ |  8jr0_validation.xml.gz 8jr0_validation.xml.gz | 127.8 KB | 表示 | |
| CIF形式データ |  8jr0_validation.cif.gz 8jr0_validation.cif.gz | 195.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jr/8jr0 https://data.pdbj.org/pub/pdb/validation_reports/jr/8jr0 ftp://data.pdbj.org/pub/pdb/validation_reports/jr/8jr0 ftp://data.pdbj.org/pub/pdb/validation_reports/jr/8jr0 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  36589MC  8j0sC  8j0tC 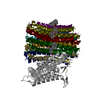 8j57C  8j58C  8jr1C  8khfC  8ki3C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP synthase ... , 7種, 19分子 ABCDEFGH123456789ab
| #1: タンパク質 | 分子量: 59358.070 Da / 分子数: 3 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpA, Rv1308, MTCY373.28 / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPU7, H+-transporting two-sector ATPase Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPU7, H+-transporting two-sector ATPase#2: タンパク質 | 分子量: 53150.934 Da / 分子数: 3 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpD, Rv1310, MTCY373.30 / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPU5, H+-transporting two-sector ATPase Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPU5, H+-transporting two-sector ATPase#3: タンパク質 | | 分子量: 33929.332 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpG, Rv1309, MTCY373.29 / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPU9 Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPU9#4: タンパク質 | | 分子量: 13149.744 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpC, Rv1311, MTCY373.31 / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPV1 Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: P9WPV1#5: タンパク質 | 分子量: 8058.423 Da / 分子数: 9 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpE / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045H4W8 Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045H4W8#6: タンパク質 | | 分子量: 27488.436 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpB, ERS007657_00358, ERS007661_00092, ERS007663_00105, ERS007665_00910, ERS007670_00031, ERS007679_03316, ERS007681_03471, ERS007688_02939, ERS007703_00159, ERS007720_03212, ERS007722_ ...遺伝子: atpB, ERS007657_00358, ERS007661_00092, ERS007663_00105, ERS007665_00910, ERS007670_00031, ERS007679_03316, ERS007681_03471, ERS007688_02939, ERS007703_00159, ERS007720_03212, ERS007722_00190, ERS007739_02359, ERS007741_03568, ERS024276_02583, ERS027646_02991, ERS027659_00329, ERS027661_00021, SAMEA2683035_01568 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045J1C5 Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045J1C5#7: タンパク質 | | 分子量: 18345.771 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpF / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045H294 Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045H294 |
|---|
-タンパク質 , 1種, 1分子 d
| #8: タンパク質 | 分子量: 48866.648 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: atpH / 発現宿主:  Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045JVE3 Mycolicibacterium smegmatis (バクテリア) / 参照: UniProt: A0A045JVE3 |
|---|
-非ポリマー , 4種, 17分子 




| #9: 化合物 | | #10: 化合物 | ChemComp-MG / #11: 化合物 | #12: 化合物 | ChemComp-UTI / ( 分子量: 614.503 Da / 分子数: 7 / 由来タイプ: 合成 / 式: C30H33BrFN3O5 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|---|
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Mycobacterium tuberculosis ATP synthase / タイプ: COMPLEX / Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2400 nm / 最小 デフォーカス(公称値): 1200 nm |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 81528 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj





