+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8bsu | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of the kainate receptor GluK3-H523A ligand binding domain in complex with kainate and the positive allosteric modulator BPAM344 at 2.9A resolution | ||||||
 要素 要素 | Glutamate receptor ionotropic, kainate 3 | ||||||
 キーワード キーワード | MEMBRANE PROTEIN / IONOTROPIC GLUTAMATE RECEPTOR / GLUK3 LIGAND-BINDING DOMAIN / GluK3-H523A-LBD / positive allosteric modulator / BPAM344 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Presynaptic function of Kainate receptors / cochlear hair cell ribbon synapse / adenylate cyclase inhibiting G protein-coupled glutamate receptor activity / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / G protein-coupled glutamate receptor signaling pathway / glutamate receptor activity / negative regulation of synaptic transmission, glutamatergic / glutamate receptor signaling pathway / kainate selective glutamate receptor activity ...Presynaptic function of Kainate receptors / cochlear hair cell ribbon synapse / adenylate cyclase inhibiting G protein-coupled glutamate receptor activity / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / G protein-coupled glutamate receptor signaling pathway / glutamate receptor activity / negative regulation of synaptic transmission, glutamatergic / glutamate receptor signaling pathway / kainate selective glutamate receptor activity / glutamate-gated receptor activity / glutamate-gated calcium ion channel activity / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / dendrite cytoplasm / regulation of membrane potential / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / synaptic transmission, glutamatergic / postsynaptic density membrane / modulation of chemical synaptic transmission / terminal bouton / presynaptic membrane / perikaryon / chemical synaptic transmission / postsynaptic membrane / axon / dendrite / glutamatergic synapse / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.9 Å 分子置換 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Venskutonyte, R. / Frydenvang, K. / Kastrup, J.S. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: FEBS J / 年: 2024 ジャーナル: FEBS J / 年: 2024タイトル: Small-molecule positive allosteric modulation of homomeric kainate receptors GluK1-3: development of screening assays and insight into GluK3 structure. 著者: Yasmin Bay / Raminta Venskutonytė / Stine M Frantsen / Thor S Thorsen / Maria Musgaard / Karla Frydenvang / Pierre Francotte / Bernard Pirotte / Philip C Biggin / Anders S Kristensen / ...著者: Yasmin Bay / Raminta Venskutonytė / Stine M Frantsen / Thor S Thorsen / Maria Musgaard / Karla Frydenvang / Pierre Francotte / Bernard Pirotte / Philip C Biggin / Anders S Kristensen / Thomas Boesen / Darryl S Pickering / Michael Gajhede / Jette S Kastrup /    要旨: The kainate receptors GluK1-3 (glutamate receptor ionotropic, kainate receptors 1-3) belong to the family of ionotropic glutamate receptors and are essential for fast excitatory neurotransmission in ...The kainate receptors GluK1-3 (glutamate receptor ionotropic, kainate receptors 1-3) belong to the family of ionotropic glutamate receptors and are essential for fast excitatory neurotransmission in the brain, and are associated with neurological and psychiatric diseases. How these receptors can be modulated by small-molecule agents is not well understood, especially for GluK3. We show that the positive allosteric modulator BPAM344 can be used to establish robust calcium-sensitive fluorescence-based assays to test agonists, antagonists, and positive allosteric modulators of GluK1-3. The half-maximal effective concentration (EC) of BPAM344 for potentiating the response of 100 μm kainate was determined to be 26.3 μm for GluK1, 75.4 μm for GluK2, and 639 μm for GluK3. Domoate was found to be a potent agonist for GluK1 and GluK2, with an EC of 0.77 and 1.33 μm, respectively, upon co-application of 150 μm BPAM344. At GluK3, domoate acts as a very weak agonist or antagonist with a half-maximal inhibitory concentration (IC) of 14.5 μm, in presence of 500 μm BPAM344 and 100 μm kainate for competition binding. Using H523A-mutated GluK3, we determined the first dimeric structure of the ligand-binding domain by X-ray crystallography, allowing location of BPAM344, as well as zinc-, sodium-, and chloride-ion binding sites at the dimer interface. Molecular dynamics simulations support the stability of the ion sites as well as the involvement of Asp761, Asp790, and Glu797 in the binding of zinc ions. Using electron microscopy, we show that, in presence of glutamate and BPAM344, full-length GluK3 adopts a dimer-of-dimers arrangement. #1:  ジャーナル: Febs J. / 年: 2024 ジャーナル: Febs J. / 年: 2024タイトル: Small-molecule positive allosteric modulation of homomeric kainate receptors GluK1-3: development of screening assays and insight into GluK3 structure. 著者: Bay, Y. / Venskutonyte, R. / Frantsen, S.M. / Thorsen, T.S. / Musgaard, M. / Frydenvang, K. / Francotte, P. / Pirotte, B. / Biggin, P.C. / Kristensen, A.S. / Boesen, T. / Pickering, D.S. / ...著者: Bay, Y. / Venskutonyte, R. / Frantsen, S.M. / Thorsen, T.S. / Musgaard, M. / Frydenvang, K. / Francotte, P. / Pirotte, B. / Biggin, P.C. / Kristensen, A.S. / Boesen, T. / Pickering, D.S. / Gajhede, M. / Kastrup, J.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8bsu.cif.gz 8bsu.cif.gz | 793.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8bsu.ent.gz pdb8bsu.ent.gz | 663.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8bsu.json.gz 8bsu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8bsu_validation.pdf.gz 8bsu_validation.pdf.gz | 5.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8bsu_full_validation.pdf.gz 8bsu_full_validation.pdf.gz | 5.3 MB | 表示 | |
| XML形式データ |  8bsu_validation.xml.gz 8bsu_validation.xml.gz | 74.7 KB | 表示 | |
| CIF形式データ |  8bsu_validation.cif.gz 8bsu_validation.cif.gz | 95.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bs/8bsu https://data.pdbj.org/pub/pdb/validation_reports/bs/8bsu ftp://data.pdbj.org/pub/pdb/validation_reports/bs/8bsu ftp://data.pdbj.org/pub/pdb/validation_reports/bs/8bsu | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bstC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 8分子 ABCDEFGH
| #1: タンパク質 | 分子量: 29025.383 Da / 分子数: 8 / 変異: H523A / 由来タイプ: 組換発現 詳細: THE PROTEIN CRYSTALLIZED IS THE EXTRACELLULAR LIGAND BINDING DOMAIN OF GLUK3-H523A. TRANSMEMBRANE REGIONS WERE GENETICALLY REMOVED AND REPLACED WITH A GLY-THR LINKER (RESIDUES 119 AND 120 OF ...詳細: THE PROTEIN CRYSTALLIZED IS THE EXTRACELLULAR LIGAND BINDING DOMAIN OF GLUK3-H523A. TRANSMEMBRANE REGIONS WERE GENETICALLY REMOVED AND REPLACED WITH A GLY-THR LINKER (RESIDUES 119 AND 120 OF THE STRUCTURE). THEREFORE, THE SEQUENCE MATCHES DISCONTINUOUSLY WITH THE REFERENCE DATABASE (432-546, 669-806) 由来: (組換発現)   |
|---|
-非ポリマー , 8種, 197分子 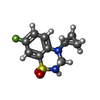














| #2: 化合物 | ChemComp-2J9 / #3: 化合物 | ChemComp-ZN / #4: 化合物 | ChemComp-GOL / #5: 化合物 | ChemComp-CL / #6: 化合物 | ChemComp-SO4 / #7: 化合物 | ChemComp-KAI / #8: 化合物 | #9: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.32 Å3/Da / 溶媒含有率: 47.01 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 詳細: PEG 4000, lithium sulfate, zinc acetate, Tris buffer pH 8.5 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  MAX II MAX II  / ビームライン: I911-3 / 波長: 1 Å / ビームライン: I911-3 / 波長: 1 Å |
| 検出器 | タイプ: MARMOSAIC 225 mm CCD / 検出器: CCD / 日付: 2015年3月29日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.9→46.84 Å / Num. obs: 47190 / % possible obs: 99.9 % / 冗長度: 4.1 % / Rpim(I) all: 0.066 / Rrim(I) all: 0.133 / Net I/σ(I): 11.6 / Num. measured all: 192159 |
| 反射 シェル | 解像度: 2.9→3.06 Å / % possible obs: 99.7 % / 冗長度: 4.1 % / Num. measured all: 27910 / Num. unique obs: 6808 / Rpim(I) all: 0.245 / Rrim(I) all: 0.498 / Net I/σ(I) obs: 3.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 2.9→45.33 Å / SU ML: 0.37 / 交差検証法: THROUGHOUT / σ(F): 1.36 / 位相誤差: 23.99 / 立体化学のターゲット値: ML 分子置換 / 解像度: 2.9→45.33 Å / SU ML: 0.37 / 交差検証法: THROUGHOUT / σ(F): 1.36 / 位相誤差: 23.99 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.9→45.33 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj








