+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8bh0 | ||||||
|---|---|---|---|---|---|---|---|
| Title | O-Methyltransferase Plu4890 in complex with SAH and AQ-270b | ||||||
 Components Components | Methyltransferase Plu4890 | ||||||
 Keywords Keywords | TRANSFERASE / methyltransferase / polyketide / anthraquinone | ||||||
| Function / homology |  Function and homology information Function and homology information | ||||||
| Biological species |  Photorhabdus laumondii subsp. laumondii TTO1 (bacteria) Photorhabdus laumondii subsp. laumondii TTO1 (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.7 Å MOLECULAR REPLACEMENT / Resolution: 1.7 Å | ||||||
 Authors Authors | Huber, E.M. / Groll, M. | ||||||
| Funding support | 1items
| ||||||
 Citation Citation |  Journal: Structure / Year: 2023 Journal: Structure / Year: 2023Title: A set of closely related methyltransferases for site-specific tailoring of anthraquinone pigments. Authors: Huber, E.M. / Kreling, L. / Heinrich, A.K. / Dunnebacke, M. / Pothig, A. / Bode, H.B. / Groll, M. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8bh0.cif.gz 8bh0.cif.gz | 1 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8bh0.ent.gz pdb8bh0.ent.gz | 881.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  8bh0.json.gz 8bh0.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  8bh0_validation.pdf.gz 8bh0_validation.pdf.gz | 4.9 MB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  8bh0_full_validation.pdf.gz 8bh0_full_validation.pdf.gz | 4.9 MB | Display | |
| Data in XML |  8bh0_validation.xml.gz 8bh0_validation.xml.gz | 92.6 KB | Display | |
| Data in CIF |  8bh0_validation.cif.gz 8bh0_validation.cif.gz | 127.4 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/bh/8bh0 https://data.pdbj.org/pub/pdb/validation_reports/bh/8bh0 ftp://data.pdbj.org/pub/pdb/validation_reports/bh/8bh0 ftp://data.pdbj.org/pub/pdb/validation_reports/bh/8bh0 | HTTPS FTP |
-Related structure data
| Related structure data |  8bgtSC  8bgxC  8bgyC  8bgzC  8bibC  8bicC  8bidC  8bieC  8bifC  8bigC  8bihC  8biiC  8bijC  8birC S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 8 molecules ABCDEFGH
| #1: Protein | Mass: 36550.527 Da / Num. of mol.: 8 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Photorhabdus laumondii subsp. laumondii TTO1 (bacteria) Photorhabdus laumondii subsp. laumondii TTO1 (bacteria)Production host:  |
|---|
-Non-polymers , 5 types, 879 molecules 
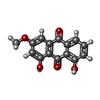







| #2: Chemical | ChemComp-SAH / #3: Chemical | ChemComp-QPO / #4: Chemical | #5: Chemical | ChemComp-NA / #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.47 Å3/Da / Density % sol: 50.11 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 6.5 Details: 0.2 M Ammoniumsulfate, 0.1 M MES pH 6.5, 30 % PEG5000MME |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SLS SLS  / Beamline: X06SA / Wavelength: 1 Å / Beamline: X06SA / Wavelength: 1 Å |
| Detector | Type: DECTRIS EIGER X 16M / Detector: PIXEL / Date: May 21, 2022 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 1.7→49 Å / Num. obs: 291332 / % possible obs: 94.8 % / Redundancy: 3.1 % / Rmerge(I) obs: 0.048 / Net I/σ(I): 11.5 |
| Reflection shell | Resolution: 1.7→1.8 Å / Rmerge(I) obs: 0.644 / Mean I/σ(I) obs: 2 / Num. unique obs: 45939 / % possible all: 94.7 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 8BGT Resolution: 1.7→30 Å / Cor.coef. Fo:Fc: 0.969 / Cor.coef. Fo:Fc free: 0.96 / SU B: 5.55 / SU ML: 0.079 / Cross valid method: THROUGHOUT / σ(F): 0 / ESU R: 0.165 / ESU R Free: 0.102 / Stereochemistry target values: MAXIMUM LIKELIHOOD Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : WITH TLS ADDED
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 76.53 Å2 / Biso mean: 30.708 Å2 / Biso min: 19.08 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.7→30 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.7→1.744 Å / Rfactor Rfree error: 0 / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller



 PDBj
PDBj



