[English] 日本語
 Yorodumi
Yorodumi- PDB-7xdj: Crystal structure of actinomycin D-echinomycin-d(AGCGCGT/ACGAGCT)... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7xdj | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
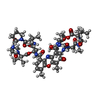
| Title | Crystal structure of actinomycin D-echinomycin-d(AGCGCGT/ACGAGCT) complex | ||||||||||||
 Components Components |
| ||||||||||||
 Keywords Keywords | DNA/ANTIBIOTIC / G:A mismatch / Single-strand DNA twist / mismatch binding antibiotic / DNA / DNA-ANTIBIOTIC complex | ||||||||||||
| Function / homology | Actinomycin D / Echinomycin / 2-CARBOXYQUINOXALINE / : / DNA Function and homology information Function and homology information | ||||||||||||
| Biological species |  Streptomyces (bacteria) Streptomyces (bacteria)synthetic construct (others) | ||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.435 Å MOLECULAR REPLACEMENT / Resolution: 2.435 Å | ||||||||||||
 Authors Authors | Chien, C.M. / Satange, R.B. / Hou, M.H. | ||||||||||||
| Funding support |  Taiwan, 2items Taiwan, 2items
| ||||||||||||
 Citation Citation |  Journal: Nucleic Acids Res. / Year: 2022 Journal: Nucleic Acids Res. / Year: 2022Title: Staggered intercalation of DNA duplexes with base-pair modulation by two distinct drug molecules induces asymmetric backbone twisting and structure polymorphism. Authors: Satange, R. / Kao, S.H. / Chien, C.M. / Chou, S.H. / Lin, C.C. / Neidle, S. / Hou, M.H. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7xdj.cif.gz 7xdj.cif.gz | 113.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7xdj.ent.gz pdb7xdj.ent.gz | 89.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7xdj.json.gz 7xdj.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/xd/7xdj https://data.pdbj.org/pub/pdb/validation_reports/xd/7xdj ftp://data.pdbj.org/pub/pdb/validation_reports/xd/7xdj ftp://data.pdbj.org/pub/pdb/validation_reports/xd/7xdj | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  7x6rC  7x97C  7x9fC  7dq0S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 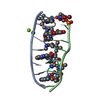
| ||||||||
| 2 | 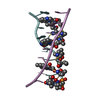
| ||||||||
| 3 | 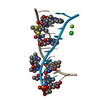
| ||||||||
| 4 | 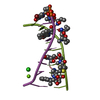
| ||||||||
| Unit cell |
|
- Components
Components
-DNA chain , 2 types, 8 molecules AEIMBFJN
| #1: DNA chain | Mass: 2138.423 Da / Num. of mol.: 4 / Source method: obtained synthetically / Source: (synth.) synthetic construct (others) #2: DNA chain | Mass: 2187.294 Da / Num. of mol.: 4 / Source method: obtained synthetically / Source: (synth.) synthetic construct (others) |
|---|
-Protein/peptide , 2 types, 8 molecules LPDHCGKO
-Non-polymers , 4 types, 48 molecules 





| #5: Chemical | | #6: Chemical | #7: Chemical | ChemComp-QUI /   Type: Cyclic depsipeptide / Class: Antibiotic / Mass: 174.156 Da / Num. of mol.: 8 / Source method: obtained synthetically / Formula: C9H6N2O2 Type: Cyclic depsipeptide / Class: Antibiotic / Mass: 174.156 Da / Num. of mol.: 8 / Source method: obtained synthetically / Formula: C9H6N2O2Details: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ...Details: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. References: Echinomycin #8: Water | ChemComp-HOH / | |
|---|
-Details
| Compound details | THE ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE, A MEMBER OF THE QUINOXALINE CLASS OF ANTIBIOTICS. ...THE ECHINOMYCI |
|---|---|
| Has ligand of interest | N |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.28 Å3/Da / Density % sol: 62.46 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, sitting drop / pH: 6.5 Details: 0.25 mM Oligonucleotide, 0.25 mM Echinomycin, 0.125 mM Actinomycin D, 2.5 mM Sodium cacodylate, 3.5 mM Magnesium chloride, 1 mM Spermine tetrahydrochloride, 6 mM Cobalt chloride, 1% (v/v) 2- ...Details: 0.25 mM Oligonucleotide, 0.25 mM Echinomycin, 0.125 mM Actinomycin D, 2.5 mM Sodium cacodylate, 3.5 mM Magnesium chloride, 1 mM Spermine tetrahydrochloride, 6 mM Cobalt chloride, 1% (v/v) 2-Methyl-2,4-pentanediol, 6% (v/v) PEG 200, Reservior PEG 200 (30%) |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSRRC NSRRC  / Beamline: BL15A1 / Wavelength: 0.91982 Å / Beamline: BL15A1 / Wavelength: 0.91982 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: RAYONIX MX300HE / Detector: CCD / Date: Apr 23, 2018 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.91982 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 2.4→30 Å / Num. obs: 24192 / % possible obs: 99.8 % / Redundancy: 11.1 % / Rmerge(I) obs: 0.084 / Rpim(I) all: 0.027 / Rrim(I) all: 0.088 / Χ2: 1.208 / Net I/σ(I): 13.6 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 7DQ0 Resolution: 2.435→24.835 Å / SU ML: 0.39 / Cross valid method: THROUGHOUT / σ(F): 1.97 / Phase error: 27.69 / Stereochemistry target values: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 130.55 Å2 / Biso mean: 57.2815 Å2 / Biso min: 36.24 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.435→24.835 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Origin x: 5.3782 Å / Origin y: 25.8104 Å / Origin z: 11.6789 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group | Selection details: all |
 Movie
Movie Controller
Controller


 PDBj
PDBj