[English] 日本語
 Yorodumi
Yorodumi- PDB-7vr8: Inward-facing structure of human EAAT2 in the substrate-free state -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7vr8 | ||||||
|---|---|---|---|---|---|---|---|
| Title | Inward-facing structure of human EAAT2 in the substrate-free state | ||||||
 Components Components | Excitatory amino acid transporter 2 | ||||||
 Keywords Keywords | TRANSPORT PROTEIN / transporter / MEMBRANE PROTEIN | ||||||
| Function / homology |  Function and homology information Function and homology informationneurotransmitter reuptake / Astrocytic Glutamate-Glutamine Uptake And Metabolism / cysteine transmembrane transporter activity / high-affinity L-glutamate transmembrane transporter activity / glutamate:sodium symporter activity / visual behavior / membrane protein complex / : / L-glutamate transmembrane transporter activity / L-glutamate transmembrane transport ...neurotransmitter reuptake / Astrocytic Glutamate-Glutamine Uptake And Metabolism / cysteine transmembrane transporter activity / high-affinity L-glutamate transmembrane transporter activity / glutamate:sodium symporter activity / visual behavior / membrane protein complex / : / L-glutamate transmembrane transporter activity / L-glutamate transmembrane transport / glutathione biosynthetic process / D-aspartate import across plasma membrane / L-aspartate transmembrane transport / monoatomic anion transmembrane transporter activity / telencephalon development / L-aspartate import across plasma membrane / Glutamate Neurotransmitter Release Cycle / L-glutamate import across plasma membrane / neutral L-amino acid transmembrane transporter activity / transepithelial transport / neuron projection terminus / cellular response to cocaine / astrocyte projection / neurotransmitter transport / protein homotrimerization / adult behavior / axolemma / response to amino acid / transport across blood-brain barrier / monoatomic ion transport / positive regulation of D-glucose import across plasma membrane / response to wounding / multicellular organism growth / presynaptic membrane / cell body / vesicle / chemical synaptic transmission / membrane raft / response to xenobiotic stimulus / glutamatergic synapse / cell surface / metal ion binding / membrane / plasma membrane Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 3.58 Å | ||||||
 Authors Authors | Kato, T. / Kusakizako, T. / Yamashita, K. / Nishizawa, T. / Nureki, O. | ||||||
| Funding support |  Japan, 1items Japan, 1items
| ||||||
 Citation Citation |  Journal: Nat Commun / Year: 2022 Journal: Nat Commun / Year: 2022Title: Structural insights into inhibitory mechanism of human excitatory amino acid transporter EAAT2. Authors: Takafumi Kato / Tsukasa Kusakizako / Chunhuan Jin / Xinyu Zhou / Ryuichi Ohgaki / LiLi Quan / Minhui Xu / Suguru Okuda / Kan Kobayashi / Keitaro Yamashita / Tomohiro Nishizawa / Yoshikatsu ...Authors: Takafumi Kato / Tsukasa Kusakizako / Chunhuan Jin / Xinyu Zhou / Ryuichi Ohgaki / LiLi Quan / Minhui Xu / Suguru Okuda / Kan Kobayashi / Keitaro Yamashita / Tomohiro Nishizawa / Yoshikatsu Kanai / Osamu Nureki /   Abstract: Glutamate is a pivotal excitatory neurotransmitter in mammalian brains, but excessive glutamate causes numerous neural disorders. Almost all extracellular glutamate is retrieved by the glial ...Glutamate is a pivotal excitatory neurotransmitter in mammalian brains, but excessive glutamate causes numerous neural disorders. Almost all extracellular glutamate is retrieved by the glial transporter, Excitatory Amino Acid Transporter 2 (EAAT2), belonging to the SLC1A family. However, in some cancers, EAAT2 expression is enhanced and causes resistance to therapies by metabolic disturbance. Despite its crucial roles, the detailed structural information about EAAT2 has not been available. Here, we report cryo-EM structures of human EAAT2 in substrate-free and selective inhibitor WAY213613-bound states at 3.2 Å and 2.8 Å, respectively. EAAT2 forms a trimer, with each protomer consisting of transport and scaffold domains. Along with a glutamate-binding site, the transport domain possesses a cavity that could be disrupted during the transport cycle. WAY213613 occupies both the glutamate-binding site and cavity of EAAT2 to interfere with its alternating access, where the sensitivity is defined by the inner environment of the cavity. We provide the characterization of the molecular features of EAAT2 and its selective inhibition mechanism that may facilitate structure-based drug design for EAAT2. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7vr8.cif.gz 7vr8.cif.gz | 100.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7vr8.ent.gz pdb7vr8.ent.gz | 69.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7vr8.json.gz 7vr8.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/vr/7vr8 https://data.pdbj.org/pub/pdb/validation_reports/vr/7vr8 ftp://data.pdbj.org/pub/pdb/validation_reports/vr/7vr8 ftp://data.pdbj.org/pub/pdb/validation_reports/vr/7vr8 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  32098MC  7vr7C M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS oper:
|
- Components
Components
| #1: Protein | Mass: 63016.879 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: SLC1A2, EAAT2, GLT1 / Production host: Homo sapiens (human) / Gene: SLC1A2, EAAT2, GLT1 / Production host:  Homo sapiens (human) / References: UniProt: P43004 Homo sapiens (human) / References: UniProt: P43004 | ||
|---|---|---|---|
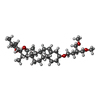
| #2: Chemical | ChemComp-9Z9 / ( | ||
| #3: Chemical | ChemComp-CLR / | ||
| #4: Chemical | | Has ligand of interest | Y | |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component | Name: EAAT2 / Type: COMPLEX / Entity ID: #1 / Source: RECOMBINANT |
|---|---|
| Molecular weight | Experimental value: NO |
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Source (recombinant) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Buffer solution | pH: 7 |
| Specimen | Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES |
| Specimen support | Grid material: COPPER/RHODIUM / Grid mesh size: 300 divisions/in. / Grid type: Quantifoil R1.2/1.3 |
| Vitrification | Cryogen name: ETHANE / Humidity: 100 % / Chamber temperature: 277 K |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: TFS KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 1600 nm / Nominal defocus min: 800 nm |
| Image recording | Electron dose: 50 e/Å2 / Film or detector model: GATAN K3 (6k x 4k) |
| EM imaging optics | Energyfilter name: GIF Bioquantum |
- Processing
Processing
| Software | Name: REFMAC / Version: 5.8.0291 / Classification: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EM software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Point symmetry: C3 (3 fold cyclic) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3D reconstruction | Resolution: 3.58 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 212554 / Symmetry type: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Atomic model building | Space: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Atomic model building | PDB-ID: 6GCT Accession code: 6GCT / Source name: PDB / Type: experimental model | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement | Resolution: 3.58→3.58 Å / Cor.coef. Fo:Fc: 0.892 / WRfactor Rwork: 0.371 / SU B: 15.602 / SU ML: 0.255 / Average fsc overall: 0.8681 / Average fsc work: 0.8681 / ESU R: 0.266 Details: Hydrogens have been added in their riding positions
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: BABINET MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 164.322 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller



 PDBj
PDBj