[English] 日本語
 Yorodumi
Yorodumi- PDB-7tce: Crystal structure of delta sub IV Rhodobacter Sphaeroides bc1 wit... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7tce | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of delta sub IV Rhodobacter Sphaeroides bc1 with the antimalarial drug atovaquone. | ||||||
 Components Components |
| ||||||
 Keywords Keywords | OXIDOREDUCTASE / Respiratory Chain Complex III / Anti-malarial Atovaquone / Rhodobacter Spaheroides bc1 / Inhibition / MEMBRANE PROTEIN | ||||||
| Function / homology |  Function and homology information Function and homology informationrespiratory chain complex III / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / respiratory electron transport chain / 2 iron, 2 sulfur cluster binding / electron transfer activity / heme binding / metal ion binding / membrane / plasma membrane Similarity search - Function | ||||||
| Biological species |  Cereibacter sphaeroides (bacteria) Cereibacter sphaeroides (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 3.85 Å MOLECULAR REPLACEMENT / Resolution: 3.85 Å | ||||||
 Authors Authors | Esser, L. / Xia, D. / Zhou, F. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Crystal structure of delta sub IV Rhodobacter Sphaeroides bc1 with the antimalarial drug atovaquone. Authors: Esser, L. / Xia, D. / Zhou, F. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7tce.cif.gz 7tce.cif.gz | 1.3 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7tce.ent.gz pdb7tce.ent.gz | 1.1 MB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7tce.json.gz 7tce.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/tc/7tce https://data.pdbj.org/pub/pdb/validation_reports/tc/7tce ftp://data.pdbj.org/pub/pdb/validation_reports/tc/7tce ftp://data.pdbj.org/pub/pdb/validation_reports/tc/7tce | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5kkzS S: Starting model for refinement |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments:
NCS ensembles :
|
- Components
Components
-Protein , 3 types, 12 molecules AEKOBFLPCGMQ
| #1: Protein | Mass: 50087.422 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Cereibacter sphaeroides (bacteria) / Gene: petB, fbcB / Production host: Cereibacter sphaeroides (bacteria) / Gene: petB, fbcB / Production host:  Cereibacter sphaeroides (bacteria) / References: UniProt: Q02761 Cereibacter sphaeroides (bacteria) / References: UniProt: Q02761#2: Protein | Mass: 29373.953 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Cereibacter sphaeroides (bacteria) / Production host: Cereibacter sphaeroides (bacteria) / Production host:  Cereibacter sphaeroides (bacteria) / References: UniProt: A3PFR5 Cereibacter sphaeroides (bacteria) / References: UniProt: A3PFR5#3: Protein | Mass: 19928.375 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Cereibacter sphaeroides (bacteria) / Gene: petA, fbcF / Production host: Cereibacter sphaeroides (bacteria) / Gene: petA, fbcF / Production host:  Cereibacter sphaeroides (bacteria) / References: UniProt: Q02762, quinol-cytochrome-c reductase Cereibacter sphaeroides (bacteria) / References: UniProt: Q02762, quinol-cytochrome-c reductase |
|---|
-Sugars , 1 types, 4 molecules 
| #8: Sugar | ChemComp-BOG / |
|---|
-Non-polymers , 6 types, 30 molecules 
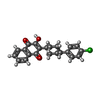









| #4: Chemical | ChemComp-HEM / #5: Chemical | ChemComp-AOQ / #6: Chemical | ChemComp-6PE / #7: Chemical | ChemComp-SR / #9: Chemical | ChemComp-HEC / #10: Chemical | ChemComp-FES / |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.52 Å3/Da / Density % sol: 65.02 % |
|---|---|
| Crystal grow | Temperature: 289 K / Method: vapor diffusion, sitting drop / pH: 8 Details: 7% PEG400, 200 MM HISTIDINE, 150 MM NACL, 10 MM SODIUM ASCORBATE, Atovaquone 5 fold molar excess, B-OG, SUCROSE MONO CAPARATE, TRIS PH 8.0. |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 22-ID / Wavelength: 1 Å / Beamline: 22-ID / Wavelength: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: DECTRIS EIGER2 S 16M / Detector: PIXEL / Date: Mar 29, 2019 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 3.85→50 Å / Num. obs: 50477 / % possible obs: 94.6 % / Redundancy: 4.8 % / Biso Wilson estimate: 152.55 Å2 / Rmerge(I) obs: 0.2 / Rpim(I) all: 0.096 / Rrim(I) all: 0.223 / Χ2: 1.007 / Net I/σ(I): 3.9 / Num. measured all: 242827 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5kkz Resolution: 3.85→39.43 Å / SU ML: 0.58 / Cross valid method: THROUGHOUT / σ(F): 0.37 / Phase error: 34.15 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 475.04 Å2 / Biso mean: 192.085 Å2 / Biso min: 108.4 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 3.85→39.43 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 10
|
 Movie
Movie Controller
Controller




 PDBj
PDBj





















