| 登録情報 | データベース: PDB / ID: 7s0p
|
|---|
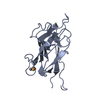
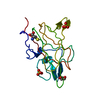
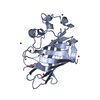
| タイトル | Crystal structure of Porcine Factor VIII C2 Domain Bound to Phosphatidylserine |
|---|
 要素 要素 | Coagulation factor VIII |
|---|
 キーワード キーワード | LIPID BINDING PROTEIN / factor VIII / lipid / hemostasis |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
blood coagulation, intrinsic pathway / acute-phase response / oxidoreductase activity / copper ion binding / extracellular space類似検索 - 分子機能 Coagulation factor 5/8-like / : / Coagulation factors 5/8 type C domain (FA58C) signature 2. / Multicopper oxidases, conserved site / Multicopper oxidases signature 1. / Coagulation factors 5/8 type C domain (FA58C) signature 1. / Coagulation factor 5/8 C-terminal domain, discoidin domain / Coagulation factors 5/8 type C domain (FA58C) profile. / Multicopper oxidase, C-terminal / Multicopper oxidase ...Coagulation factor 5/8-like / : / Coagulation factors 5/8 type C domain (FA58C) signature 2. / Multicopper oxidases, conserved site / Multicopper oxidases signature 1. / Coagulation factors 5/8 type C domain (FA58C) signature 1. / Coagulation factor 5/8 C-terminal domain, discoidin domain / Coagulation factors 5/8 type C domain (FA58C) profile. / Multicopper oxidase, C-terminal / Multicopper oxidase / F5/8 type C domain / Coagulation factor 5/8 C-terminal domain / Multicopper oxidase, type 1 / Multicopper oxidase / Multicopper oxidase, N-terminal / Multicopper oxidase / Cupredoxin / Galactose-binding-like domain superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Sus scrofa (ブタ) Sus scrofa (ブタ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.3 Å 分子置換 / 解像度: 1.3 Å |
|---|
 データ登録者 データ登録者 | Peters, S.C. / Childers, K.C. / Wo, S.W. / Brison, C.M. / Swanson, C.D. / Spiegel, P.C. |
|---|
| 資金援助 |  米国, 6件 米国, 6件 | 組織 | 認可番号 | 国 |
|---|
| National Science Foundation (NSF, United States) | MRI 1429164 |  米国 米国 | | National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | R15HL103518 |  米国 米国 | | National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | U54HL141981 |  米国 米国 | | National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | R44HL117511 |  米国 米国 | | National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | R44HL110448 |  米国 米国 | | National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | U54HL112309 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Front Mol Biosci / 年: 2022 ジャーナル: Front Mol Biosci / 年: 2022
タイトル: Stable binding to phosphatidylserine-containing membranes requires conserved arginine residues in tandem C domains of blood coagulation factor VIII.
著者: Peters, S.C. / Childers, K.C. / Mitchell, C.E. / Avery, N.G. / Reese Jr., S.S. / Mitchell, C. / Wo, S.W. / Swanson, C.D. / Brison, C.M. / Spiegel Jr., P.C. |
|---|
| 履歴 | | 登録 | 2021年8月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2021年10月27日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年11月9日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2022年11月30日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.name |
|---|
| 改定 1.3 | 2023年10月25日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.3 Å
分子置換 / 解像度: 1.3 Å  データ登録者
データ登録者 米国, 6件
米国, 6件  引用
引用 ジャーナル: Front Mol Biosci / 年: 2022
ジャーナル: Front Mol Biosci / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7s0p.cif.gz
7s0p.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7s0p.ent.gz
pdb7s0p.ent.gz PDB形式
PDB形式 7s0p.json.gz
7s0p.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/s0/7s0p
https://data.pdbj.org/pub/pdb/validation_reports/s0/7s0p ftp://data.pdbj.org/pub/pdb/validation_reports/s0/7s0p
ftp://data.pdbj.org/pub/pdb/validation_reports/s0/7s0p
 リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.1 / 波長: 1.1158 Å
/ ビームライン: 5.0.1 / 波長: 1.1158 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj