| 登録情報 | データベース: PDB / ID: 7run
|
|---|
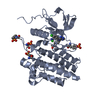
| タイトル | Crystal structure of phosphorylated RET tyrosine kinase domain complexed with a pyrrolo[2,3-d]pyrimidine inhibitor. |
|---|
 要素 要素 | Proto-oncogene tyrosine-protein kinase receptor Ret |
|---|
 キーワード キーワード | TRANSFERASE/TRANSFERASE INHIBITOR / tyrosine-protein kinase / inhibitor / cell adhesion / TRANSFERASE-TRANSFERASE INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Peyer's patch morphogenesis / GDF15-GFRAL signaling pathway / positive regulation of metanephric glomerulus development / ureter maturation / embryonic epithelial tube formation / glial cell-derived neurotrophic factor receptor signaling pathway / lymphocyte migration into lymphoid organs / posterior midgut development / membrane protein proteolysis / Formation of the ureteric bud ...Peyer's patch morphogenesis / GDF15-GFRAL signaling pathway / positive regulation of metanephric glomerulus development / ureter maturation / embryonic epithelial tube formation / glial cell-derived neurotrophic factor receptor signaling pathway / lymphocyte migration into lymphoid organs / posterior midgut development / membrane protein proteolysis / Formation of the ureteric bud / positive regulation of neuron maturation / neuron cell-cell adhesion / Formation of the nephric duct / enteric nervous system development / plasma membrane protein complex / innervation / neuron maturation / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / positive regulation of cell adhesion mediated by integrin / neural crest cell migration / ureteric bud development / regulation of axonogenesis / response to pain / homophilic cell adhesion via plasma membrane adhesion molecules / RET signaling / positive regulation of cell size / regulation of cell adhesion / cellular response to retinoic acid / transmembrane receptor protein tyrosine kinase activity / NPAS4 regulates expression of target genes / axon guidance / cell surface receptor protein tyrosine kinase signaling pathway / placental growth factor receptor activity / insulin receptor activity / vascular endothelial growth factor receptor activity / hepatocyte growth factor receptor activity / macrophage colony-stimulating factor receptor activity / platelet-derived growth factor alpha-receptor activity / platelet-derived growth factor beta-receptor activity / stem cell factor receptor activity / boss receptor activity / protein tyrosine kinase collagen receptor activity / brain-derived neurotrophic factor receptor activity / transmembrane-ephrin receptor activity / GPI-linked ephrin receptor activity / epidermal growth factor receptor activity / fibroblast growth factor receptor activity / insulin-like growth factor receptor activity / receptor protein-tyrosine kinase / positive regulation of neuron projection development / MAPK cascade / signaling receptor activity / retina development in camera-type eye / RAF/MAP kinase cascade / protein tyrosine kinase activity / early endosome / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / receptor complex / positive regulation of MAPK cascade / endosome membrane / positive regulation of cell migration / protein phosphorylation / response to xenobiotic stimulus / axon / neuronal cell body / dendrite / calcium ion binding / positive regulation of gene expression / positive regulation of DNA-templated transcription / signal transduction / ATP binding / plasma membrane類似検索 - 分子機能 Tyrosine-protein kinase, Ret receptor / Tyrosine-protein kinase receptor Ret, cadherin like domain 3 / Ret, cadherin like domain 1 / RET, cadherin-like domain 4 / : / RET Cadherin like domain 1 / RET Cadherin like domain 3 / RET Cadherin like domain 4 / RET, Cysteine Rich Domain / Cadherin domain ...Tyrosine-protein kinase, Ret receptor / Tyrosine-protein kinase receptor Ret, cadherin like domain 3 / Ret, cadherin like domain 1 / RET, cadherin-like domain 4 / : / RET Cadherin like domain 1 / RET Cadherin like domain 3 / RET Cadherin like domain 4 / RET, Cysteine Rich Domain / Cadherin domain / Cadherin-like / Cadherins domain profile. / Cadherin-like superfamily / : / Tyrosine-protein kinase, catalytic domain / Tyrosine kinase, catalytic domain / Tyrosine protein kinases specific active-site signature. / Tyrosine-protein kinase, active site / Serine-threonine/tyrosine-protein kinase, catalytic domain / Protein tyrosine and serine/threonine kinase / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily類似検索 - ドメイン・相同性 Chem-7QU / Proto-oncogene tyrosine-protein kinase receptor Ret類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.51 Å 分子置換 / 解像度: 3.51 Å |
|---|
 データ登録者 データ登録者 | Lee, C.C. / Spraggon, G. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| Not funded | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Acs Med.Chem.Lett. / 年: 2021 ジャーナル: Acs Med.Chem.Lett. / 年: 2021
タイトル: Antitarget Selectivity and Tolerability of Novel Pyrrolo[2,3- d ]pyrimidine RET Inhibitors.
著者: Mathison, C.J.N. / Yang, Y. / Nelson, J. / Huang, Z. / Jiang, J. / Chianelli, D. / Rucker, P.V. / Roland, J. / Xie, Y.F. / Epple, R. / Bursulaya, B. / Lee, C. / Gao, M.Y. / Shaffer, J. / ...著者: Mathison, C.J.N. / Yang, Y. / Nelson, J. / Huang, Z. / Jiang, J. / Chianelli, D. / Rucker, P.V. / Roland, J. / Xie, Y.F. / Epple, R. / Bursulaya, B. / Lee, C. / Gao, M.Y. / Shaffer, J. / Briones, S. / Sarkisova, Y. / Galkin, A. / Li, L. / Li, N. / Li, C. / Hua, S. / Kasibhatla, S. / Kinyamu-Akunda, J. / Kikkawa, R. / Molteni, V. / Tellew, J.E. |
|---|
| 履歴 | | 登録 | 2021年8月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2022年1月19日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年10月18日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
| 改定 1.2 | 2023年11月15日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond / Item: _chem_comp_atom.atom_id / _chem_comp_bond.atom_id_2 |
|---|
| 改定 1.3 | 2024年11月6日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3.51 Å
分子置換 / 解像度: 3.51 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Acs Med.Chem.Lett. / 年: 2021
ジャーナル: Acs Med.Chem.Lett. / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7run.cif.gz
7run.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7run.ent.gz
pdb7run.ent.gz PDB形式
PDB形式 7run.json.gz
7run.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ru/7run
https://data.pdbj.org/pub/pdb/validation_reports/ru/7run ftp://data.pdbj.org/pub/pdb/validation_reports/ru/7run
ftp://data.pdbj.org/pub/pdb/validation_reports/ru/7run
 リンク
リンク 集合体
集合体

 要素
要素 Homo sapiens (ヒト) / 遺伝子: RET, CDHF12, CDHR16, PTC, RET51
Homo sapiens (ヒト) / 遺伝子: RET, CDHF12, CDHR16, PTC, RET51
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.3 / 波長: 0.97648 Å
/ ビームライン: 5.0.3 / 波長: 0.97648 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj