+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7pz8 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Structure of an LPMO at 3.12x10^6 Gy | |||||||||
 Components Components | Gh61 isozyme a | |||||||||
 Keywords Keywords | OXIDOREDUCTASE / Lytic polysaccharide monooxygenase / metalloenzyme / copper / Auxiliary activity | |||||||||
| Function / homology |  Function and homology information Function and homology informationlytic cellulose monooxygenase (C4-dehydrogenating) / cellulase activity / cellulose catabolic process / monooxygenase activity / extracellular region / metal ion binding Similarity search - Function | |||||||||
| Biological species |  Thermoascus aurantiacus (fungus) Thermoascus aurantiacus (fungus) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.4 Å MOLECULAR REPLACEMENT / Resolution: 1.4 Å | |||||||||
 Authors Authors | Tandrup, T. / Muderspach, S.J. / Ipsen, J.O. / Johansen, K.S. / Lo Leggio, L. | |||||||||
| Funding support |  Denmark, 2items Denmark, 2items
| |||||||||
 Citation Citation |  Journal: Iucrj / Year: 2022 Journal: Iucrj / Year: 2022Title: Changes in active-site geometry on X-ray photoreduction of a lytic polysaccharide monooxygenase active-site copper and saccharide binding. Authors: Tandrup, T. / Muderspach, S.J. / Banerjee, S. / Santoni, G. / Ipsen, J.O. / Hernandez-Rollan, C. / Norholm, M.H.H. / Johansen, K.S. / Meilleur, F. / Lo Leggio, L. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7pz8.cif.gz 7pz8.cif.gz | 66.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7pz8.ent.gz pdb7pz8.ent.gz | 46.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7pz8.json.gz 7pz8.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/pz/7pz8 https://data.pdbj.org/pub/pdb/validation_reports/pz/7pz8 ftp://data.pdbj.org/pub/pdb/validation_reports/pz/7pz8 ftp://data.pdbj.org/pub/pdb/validation_reports/pz/7pz8 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  7pqrC  7pxiC  7pxjC  7pxkC  7pxlC  7pxmC  7pxnC  7pxrC  7pxsC  7pxtC  7pxuC  7pxvC  7pxwC  7pydC  7pyeC  7pyfC  7pygC  7pyhC  7pyiC  7pylC  7pymC  7pynC  7pyoC  7pypC  7pyqC  7pyuC  7pywC  7pyxC  7pyyC  7pyzC  7pz0C  7pz3C  7pz4C  7pz5C  7pz6C  7pz7C 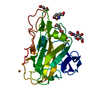 3zudS C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein / Sugars , 2 types, 2 molecules A

| #1: Protein | Mass: 24418.043 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Thermoascus aurantiacus (fungus) / Production host: Thermoascus aurantiacus (fungus) / Production host:  |
|---|---|
| #4: Sugar | ChemComp-NAG / |
-Non-polymers , 4 types, 278 molecules 





| #2: Chemical | ChemComp-CU / |
|---|---|
| #3: Chemical | ChemComp-AKR / |
| #5: Chemical | ChemComp-EPE / |
| #6: Water | ChemComp-HOH / |
-Details
| Has ligand of interest | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.22 Å3/Da / Density % sol: 44.62 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 7.5 Details: 20 mM MgCl2, 0.1 M HEPES pH 7.5, 22 %(w/v) Polyacrylic acid 5100 sodium salt. |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  PETRA III, DESY PETRA III, DESY  / Beamline: P11 / Wavelength: 1.033 Å / Beamline: P11 / Wavelength: 1.033 Å |
| Detector | Type: DECTRIS EIGER2 X 16M / Detector: PIXEL / Date: Sep 3, 2020 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.033 Å / Relative weight: 1 |
| Reflection | Resolution: 1.4→50 Å / Num. obs: 75532 / % possible obs: 91.1 % / Redundancy: 1.53 % / CC1/2: 0.998 / Net I/σ(I): 8.83 |
| Reflection shell | Resolution: 1.4→1.44 Å / Num. unique obs: 5685 / CC1/2: 0.74 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3ZUD Resolution: 1.4→43.63 Å / Cor.coef. Fo:Fc: 0.976 / Cor.coef. Fo:Fc free: 0.972 / Cross valid method: THROUGHOUT / σ(F): 0 / Stereochemistry target values: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||
| Solvent computation | Solvent model: MASK | ||||||||||||||||||||||||
| Displacement parameters | Biso max: 54.82 Å2 / Biso mean: 14.374 Å2 / Biso min: 8.57 Å2 | ||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.4→43.63 Å
|
 Movie
Movie Controller
Controller



 PDBj
PDBj



