| 登録情報 | データベース: PDB / ID: 7p08
|
|---|
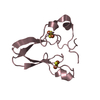
| タイトル | Solution structure of the human SF3A1 ubiquitin-like domain |
|---|
 要素 要素 | Isoform 1 of Splicing factor 3A subunit 1 |
|---|
 キーワード キーワード | SPLICING / Spliceosome / U2 snRNP / SF3A / ubiqutin-like domain |
|---|
| 機能・相同性 | Isoform 1 of Splicing factor 3A subunit 1 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | de Vries, T. / Campagne, S. / Allain, F.H.T. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2022 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2022
タイトル: Sequence-specific RNA recognition by an RGG motif connects U1 and U2 snRNP for spliceosome assembly.
著者: de Vries, T. / Martelly, W. / Campagne, S. / Sabath, K. / Sarnowski, C.P. / Wong, J. / Leitner, A. / Jonas, S. / Sharma, S. / Allain, F.H. |
|---|
| 履歴 | | 登録 | 2021年6月29日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2022年2月9日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年2月16日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.name |
|---|
| 改定 1.2 | 2024年6月19日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2022
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7p08.cif.gz
7p08.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7p08.ent.gz
pdb7p08.ent.gz PDB形式
PDB形式 7p08.json.gz
7p08.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7p08_validation.pdf.gz
7p08_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7p08_full_validation.pdf.gz
7p08_full_validation.pdf.gz 7p08_validation.xml.gz
7p08_validation.xml.gz 7p08_validation.cif.gz
7p08_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/p0/7p08
https://data.pdbj.org/pub/pdb/validation_reports/p0/7p08 ftp://data.pdbj.org/pub/pdb/validation_reports/p0/7p08
ftp://data.pdbj.org/pub/pdb/validation_reports/p0/7p08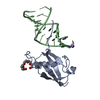
 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: SF3A1, SAP114 / プラスミド: pET24b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SF3A1, SAP114 / プラスミド: pET24b / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj
 HSQC
HSQC