[English] 日本語
 Yorodumi
Yorodumi- PDB-7ors: Ternary complex of 14-3-3 sigma, p27pT198 phosphopeptide, and WQ147 -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7ors | ||||||
|---|---|---|---|---|---|---|---|
| Title | Ternary complex of 14-3-3 sigma, p27pT198 phosphopeptide, and WQ147 | ||||||
 Components Components |
| ||||||
 Keywords Keywords | SIGNALING PROTEIN / protein-peptide complex small-molecule | ||||||
| Function / homology |  Function and homology information Function and homology informationcyclin-dependent protein kinase regulator activity / regulation of lens fiber cell differentiation / negative regulation of cyclin-dependent protein kinase activity / negative regulation of cardiac muscle tissue regeneration / negative regulation of kinase activity / autophagic cell death / FOXO-mediated transcription of cell cycle genes / negative regulation of epithelial cell proliferation involved in prostate gland development / negative regulation of cyclin-dependent protein serine/threonine kinase activity / regulation of cell cycle G1/S phase transition ...cyclin-dependent protein kinase regulator activity / regulation of lens fiber cell differentiation / negative regulation of cyclin-dependent protein kinase activity / negative regulation of cardiac muscle tissue regeneration / negative regulation of kinase activity / autophagic cell death / FOXO-mediated transcription of cell cycle genes / negative regulation of epithelial cell proliferation involved in prostate gland development / negative regulation of cyclin-dependent protein serine/threonine kinase activity / regulation of cell cycle G1/S phase transition / regulation of exit from mitosis / negative regulation of epithelial cell apoptotic process / epithelial cell proliferation involved in prostate gland development / cyclin-dependent protein serine/threonine kinase inhibitor activity / ubiquitin ligase activator activity / regulation of cyclin-dependent protein serine/threonine kinase activity / RHO GTPases activate CIT / nuclear export / negative regulation of mitotic cell cycle / AKT phosphorylates targets in the cytosol / epithelial cell apoptotic process / cellular response to antibiotic / molecular function inhibitor activity / cellular response to lithium ion / Cul4A-RING E3 ubiquitin ligase complex / regulation of epidermal cell division / protein kinase C inhibitor activity / positive regulation of epidermal cell differentiation / keratinocyte development / keratinization / p53-Dependent G1 DNA Damage Response / PTK6 Regulates Cell Cycle / Constitutive Signaling by AKT1 E17K in Cancer / regulation of cell-cell adhesion / protein kinase inhibitor activity / Defective binding of RB1 mutants to E2F1,(E2F2, E2F3) / inner ear development / negative regulation of vascular associated smooth muscle cell proliferation / regulation of G1/S transition of mitotic cell cycle / cAMP/PKA signal transduction / Regulation of localization of FOXO transcription factors / keratinocyte proliferation / Activation of BAD and translocation to mitochondria / Estrogen-dependent nuclear events downstream of ESR-membrane signaling / phosphoserine residue binding / negative regulation of keratinocyte proliferation / establishment of skin barrier / negative regulation of protein localization to plasma membrane / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest / Chk1/Chk2(Cds1) mediated inactivation of Cyclin B:Cdk1 complex / SARS-CoV-2 targets host intracellular signalling and regulatory pathways / Cyclin E associated events during G1/S transition / negative regulation of protein kinase activity / negative regulation of stem cell proliferation / Cyclin A:Cdk2-associated events at S phase entry / RHO GTPases activate PKNs / SARS-CoV-1 targets host intracellular signalling and regulatory pathways / positive regulation of protein localization / Notch signaling pathway / FLT3 Signaling / positive regulation of microtubule polymerization / positive regulation of cell adhesion / protein sequestering activity / negative regulation of innate immune response / regulation of cell migration / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / cyclin binding / protein export from nucleus / release of cytochrome c from mitochondria / placenta development / positive regulation of DNA replication / positive regulation of protein export from nucleus / stem cell proliferation / TP53 Regulates Metabolic Genes / Translocation of SLC2A4 (GLUT4) to the plasma membrane / DNA damage response, signal transduction by p53 class mediator / sensory perception of sound / G1/S transition of mitotic cell cycle / negative regulation of cell growth / potassium ion transport / DNA Damage/Telomere Stress Induced Senescence / SCF(Skp2)-mediated degradation of p27/p21 / intrinsic apoptotic signaling pathway in response to DNA damage / negative regulation of epithelial cell proliferation / Cyclin D associated events in G1 / positive regulation of protein catabolic process / cellular senescence / intracellular protein localization / protein-folding chaperone binding / regulation of protein localization / heart development / positive regulation of cell growth / Senescence-Associated Secretory Phenotype (SASP) / protein phosphatase binding / molecular adaptor activity / endosome / regulation of cell cycle / cilium / ciliary basal body / cadherin binding Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / Resolution: 1.8 Å MOLECULAR REPLACEMENT / Resolution: 1.8 Å | ||||||
 Authors Authors | Centorrino, F. / Wu, Q. / Ottmann, C. | ||||||
| Funding support | European Union, 1items
| ||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: A crystallography-based study of fragment extensions into the 14-3-3 binding groove Authors: Centorrino, F. / Wu, Q. / Cossar, P. / Brunsveld, L. / Ottmann, C. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7ors.cif.gz 7ors.cif.gz | 140.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7ors.ent.gz pdb7ors.ent.gz | 88.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7ors.json.gz 7ors.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  7ors_validation.pdf.gz 7ors_validation.pdf.gz | 752.2 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  7ors_full_validation.pdf.gz 7ors_full_validation.pdf.gz | 753.8 KB | Display | |
| Data in XML |  7ors_validation.xml.gz 7ors_validation.xml.gz | 15.4 KB | Display | |
| Data in CIF |  7ors_validation.cif.gz 7ors_validation.cif.gz | 24 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/or/7ors https://data.pdbj.org/pub/pdb/validation_reports/or/7ors ftp://data.pdbj.org/pub/pdb/validation_reports/or/7ors ftp://data.pdbj.org/pub/pdb/validation_reports/or/7ors | HTTPS FTP |
-Related structure data
| Related structure data |  7opwC  7oq7C  7oq8C  7oq9C  7oqaC  7oqgC  7oqjC  7oqsC  7oquC  7oqwC  7or3C  7or5C  7or7C  7or8C  7orgC  7orhC  7ortC  4jc3S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| Unit cell |
| ||||||||||||
| Components on special symmetry positions |
|
- Components
Components
-Protein / Protein/peptide , 2 types, 2 molecules AP
| #1: Protein | Mass: 28226.518 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: SFN, HME1 / Production host: Homo sapiens (human) / Gene: SFN, HME1 / Production host:  |
|---|---|
| #2: Protein/peptide | Mass: 1522.712 Da / Num. of mol.: 1 / Source method: obtained synthetically / Source: (synth.)  Homo sapiens (human) / References: UniProt: P46527 Homo sapiens (human) / References: UniProt: P46527 |
-Non-polymers , 4 types, 379 molecules 

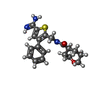




| #3: Chemical | | #4: Chemical | ChemComp-CL / | #5: Chemical | ChemComp-0I2 / ( | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.43 Å3/Da / Density % sol: 49.48 % |
|---|---|
| Crystal grow | Temperature: 277.15 K / Method: vapor diffusion, sitting drop Details: 0.095 M Hepes pH7.3, 26%PEG 400, 0.19 M CaCl2, and 5 % Glycerol |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source: SEALED TUBE / Type: RIGAKU MICROMAX-003 / Wavelength: 1.54187 Å |
| Detector | Type: DECTRIS PILATUS 200K / Detector: PIXEL / Date: Feb 22, 2021 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.54187 Å / Relative weight: 1 |
| Reflection | Resolution: 1.8→41.74 Å / Num. obs: 26844 / % possible obs: 98.5 % / Redundancy: 6.2 % / Biso Wilson estimate: 10.49 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.061 / Net I/σ(I): 19 |
| Reflection shell | Resolution: 1.8→1.83 Å / Redundancy: 5.1 % / Rmerge(I) obs: 0.188 / Mean I/σ(I) obs: 6.3 / Num. unique obs: 1252 / CC1/2: 0.974 / % possible all: 93.4 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4jc3 Resolution: 1.8→41.74 Å / SU ML: 0.1476 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 16.9916 / Stereochemistry target values: GeoStd + Monomer Library
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 14.01 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.8→41.74 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller


 PDBj
PDBj





















