ソフトウェア 名称 バージョン 分類 5.8.0267精密化 データ削減 データスケーリング 位相決定
精密化 構造決定の手法 開始モデル 解像度 / Cor.coef. Fo :Fc / Cor.coef. Fo :Fc free / SU B / SU ML / 交差検証法 / ESU R / ESU R Free / 詳細 Rfactor 反射数 %反射 Rfree 0.263 298 5.383 % Rwork 0.2037 5238 - all 0.207 - - obs - 5536 99.982 %
溶媒の処理 イオンプローブ半径 / 減衰半径 / VDWプローブ半径 / 溶媒モデル 原子変位パラメータ Biso mean 2 Baniso -1 Baniso -2 Baniso -3 1- 0.012 Å2 0.006 Å2 0 Å2 2- - 0.012 Å2 -0 Å2 3- - - -0.039 Å2
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 0 378 327 47 752
拘束条件 大きな表を表示 (5 x 23) 大きな表を隠す Refine-ID タイプ Dev ideal Dev ideal target 数 X-RAY DIFFRACTION r_bond_refined_d0.027 0.018 788 X-RAY DIFFRACTION r_bond_other_d0.028 0.025 350 X-RAY DIFFRACTION r_angle_refined_deg3.366 2.144 1196 X-RAY DIFFRACTION r_angle_other_deg3.786 3.208 822 X-RAY DIFFRACTION r_chiral_restr0.52 0.2 142 X-RAY DIFFRACTION r_chiral_restr_other1.733 0.2 22 X-RAY DIFFRACTION r_gen_planes_refined0.014 0.021 400 X-RAY DIFFRACTION r_gen_planes_other0.001 0.023 148 X-RAY DIFFRACTION r_nbd_refined0.112 0.2 94 X-RAY DIFFRACTION r_symmetry_nbd_other0.221 0.2 482 X-RAY DIFFRACTION r_nbtor_refined0.246 0.2 317 X-RAY DIFFRACTION r_symmetry_nbtor_other0.277 0.2 170 X-RAY DIFFRACTION r_xyhbond_nbd_refined0.222 0.2 69 X-RAY DIFFRACTION r_symmetry_xyhbond_nbd_other0.038 0.2 2 X-RAY DIFFRACTION r_symmetry_nbd_refined0.144 0.2 14 X-RAY DIFFRACTION r_nbd_other0.177 0.2 37 X-RAY DIFFRACTION r_symmetry_xyhbond_nbd_refined0.262 0.2 16 X-RAY DIFFRACTION r_scbond_it2.296 2.915 788 X-RAY DIFFRACTION r_scbond_other2.294 2.914 789 X-RAY DIFFRACTION r_scangle_it3.247 4.416 1196 X-RAY DIFFRACTION r_scangle_other3.246 4.415 1197 X-RAY DIFFRACTION r_lrange_it4.638 26.805 1116 X-RAY DIFFRACTION r_lrange_other4.529 26.661 1102
LS精密化 シェル 大きな表を表示 (7 x 20) 大きな表を隠す 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Refine-ID % reflection obs (%)2.281-2.34 0.253 23 0.232 378 X-RAY DIFFRACTION 100 2.34-2.404 0.221 20 0.203 373 X-RAY DIFFRACTION 100 2.404-2.474 0.237 12 0.181 365 X-RAY DIFFRACTION 100 2.474-2.55 0.271 9 0.159 365 X-RAY DIFFRACTION 100 2.55-2.633 0.129 12 333 X-RAY DIFFRACTION 100 2.633-2.725 0.421 23 0.253 325 X-RAY DIFFRACTION 100 2.725-2.828 0.23 22 309 X-RAY DIFFRACTION 100 2.828-2.944 0.299 22 0.219 299 X-RAY DIFFRACTION 100 2.944-3.074 0.22 6 308 X-RAY DIFFRACTION 100 3.074-3.224 0.244 19 0.224 264 X-RAY DIFFRACTION 100 3.224-3.398 0.281 16 0.2 284 X-RAY DIFFRACTION 100 3.398-3.604 0.311 13 0.196 254 X-RAY DIFFRACTION 100 3.604-3.852 0.341 12 0.194 245 X-RAY DIFFRACTION 100 3.852-4.16 0.3 18 0.182 221 X-RAY DIFFRACTION 100 4.16-4.556 0.136 14 0.135 207 X-RAY DIFFRACTION 100 4.556-5.092 0.264 16 0.177 191 X-RAY DIFFRACTION 100 5.092-5.876 0.236 11 0.153 174 X-RAY DIFFRACTION 100 5.876-7.187 0.246 19 0.223 138 X-RAY DIFFRACTION 100 7.187-10.124 0.444 9 0.256 119 X-RAY DIFFRACTION 100 10.124-50 0.439 2 0.281 86 X-RAY DIFFRACTION 100
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.281 Å
分子置換 / 解像度: 2.281 Å  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2021
ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7kuo.cif.gz
7kuo.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7kuo.ent.gz
pdb7kuo.ent.gz PDB形式
PDB形式 7kuo.json.gz
7kuo.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ku/7kuo
https://data.pdbj.org/pub/pdb/validation_reports/ku/7kuo ftp://data.pdbj.org/pub/pdb/validation_reports/ku/7kuo
ftp://data.pdbj.org/pub/pdb/validation_reports/ku/7kuo リンク
リンク 集合体
集合体
 要素
要素 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.1 / 波長: 0.977408 Å
/ ビームライン: 5.0.1 / 波長: 0.977408 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー







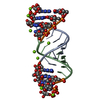









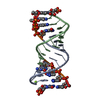
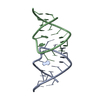



 PDBj
PDBj