[English] 日本語
 Yorodumi
Yorodumi- PDB-7kul: DNA modification at 3' end of RNA primer complex with guanosine d... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7kul | |||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
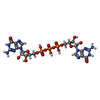
| Title | DNA modification at 3' end of RNA primer complex with guanosine dinucleotide ligand G(5')ppp(5')G | |||||||||||||||||||||||||||||||
 Components Components | DNA/RNA (5'-R(* Keywords KeywordsRNA / nonenzymatic RNA extension / DNA | Function / homology | DIGUANOSINE-5'-TRIPHOSPHATE / DNA/RNA hybrid / DNA/RNA hybrid (> 10) |  Function and homology information Function and homology informationBiological species | synthetic construct (others) | Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.64 Å MOLECULAR REPLACEMENT / Resolution: 1.64 Å  Authors AuthorsFang, Z. / Giurgiu, C. / Szostak, J.W. | Funding support | |  United States, 2items United States, 2items
 Citation Citation Journal: Angew.Chem.Int.Ed.Engl. / Year: 2021 Journal: Angew.Chem.Int.Ed.Engl. / Year: 2021Title: Structure-Activity Relationships in Nonenzymatic Template-Directed RNA Synthesis. Authors: Giurgiu, C. / Fang, Z. / Aitken, H.R.M. / Kim, S.C. / Pazienza, L. / Mittal, S. / Szostak, J.W. History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7kul.cif.gz 7kul.cif.gz | 38.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7kul.ent.gz pdb7kul.ent.gz | 25.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7kul.json.gz 7kul.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ku/7kul https://data.pdbj.org/pub/pdb/validation_reports/ku/7kul ftp://data.pdbj.org/pub/pdb/validation_reports/ku/7kul ftp://data.pdbj.org/pub/pdb/validation_reports/ku/7kul | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  7kukC  7kumC  7kunC  7kuoC  7kupC  7lneC  7lnfC  7lngC  5ueeS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| Unit cell |
| |||||||||
| Components on special symmetry positions |
|
- Components
Components
| #1: DNA/RNA hybrid | Mass: 4416.824 Da / Num. of mol.: 2 / Source method: obtained synthetically / Source: (synth.) synthetic construct (others) #2: Chemical | #3: Chemical | ChemComp-MG / #4: Water | ChemComp-HOH / | Has ligand of interest | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.05 Å3/Da / Density % sol: 54.49 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 6.5 Details: 0.2 M Magnesium acetate tetrahydrate, 0.1 M Sodium cacodylate trihydrate pH 6.5, 30% v/v (+/-)-2-Methyl-2,4-pentanediol, 50 mM Magnesium Chloride |
-Data collection
| Diffraction | Mean temperature: 99 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 8.2.1 / Wavelength: 1 Å / Beamline: 8.2.1 / Wavelength: 1 Å |
| Detector | Type: ADSC QUANTUM 315r / Detector: CCD / Date: Oct 1, 2020 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 1.64→50 Å / Num. obs: 13719 / % possible obs: 99 % / Redundancy: 9.6 % / CC1/2: 0.985 / CC star: 0.996 / Rmerge(I) obs: 0.075 / Rpim(I) all: 0.026 / Rrim(I) all: 0.079 / Χ2: 0.953 / Net I/σ(I): 24.9 |
| Reflection shell | Resolution: 1.64→1.67 Å / Redundancy: 5.4 % / Rmerge(I) obs: 0.314 / Mean I/σ(I) obs: 5.3 / Num. unique obs: 643 / CC1/2: 0.989 / CC star: 0.997 / Rpim(I) all: 0.123 / Rrim(I) all: 0.339 / Χ2: 0.903 / % possible all: 97.4 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5UEE Resolution: 1.64→50 Å / Cor.coef. Fo:Fc: 0.943 / Cor.coef. Fo:Fc free: 0.924 / SU B: 1.864 / SU ML: 0.064 / Cross valid method: THROUGHOUT / ESU R: 0.114 / ESU R Free: 0.115 Details: Hydrogens have been added in their riding positions
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK BULK SOLVENT | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 18.93 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.64→50 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller














 PDBj
PDBj