| 登録情報 | データベース: PDB / ID: 6yyp
|
|---|
| タイトル | Structure of Cathepsin S in complex with Compound 2 |
|---|
 要素 要素 | Cathepsin S |
|---|
 キーワード キーワード | HYDROLASE / Cathepsin S / inhibitor |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
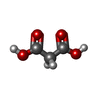
cathepsin S / regulation of antigen processing and presentation / basement membrane disassembly / positive regulation of cation channel activity / antigen processing and presentation of peptide antigen / endolysosome lumen / response to acidic pH / cellular response to thyroid hormone stimulus / Trafficking and processing of endosomal TLR / proteoglycan binding ...cathepsin S / regulation of antigen processing and presentation / basement membrane disassembly / positive regulation of cation channel activity / antigen processing and presentation of peptide antigen / endolysosome lumen / response to acidic pH / cellular response to thyroid hormone stimulus / Trafficking and processing of endosomal TLR / proteoglycan binding / Assembly of collagen fibrils and other multimeric structures / toll-like receptor signaling pathway / antigen processing and presentation / collagen catabolic process / fibronectin binding / extracellular matrix disassembly / collagen binding / laminin binding / phagocytic vesicle / Degradation of the extracellular matrix / MHC class II antigen presentation / cysteine-type peptidase activity / lysosomal lumen / proteolysis involved in protein catabolic process / Endosomal/Vacuolar pathway / antigen processing and presentation of exogenous peptide antigen via MHC class II / protein processing / tertiary granule lumen / late endosome / : / adaptive immune response / ficolin-1-rich granule lumen / lysosome / immune response / serine-type endopeptidase activity / cysteine-type endopeptidase activity / intracellular membrane-bounded organelle / Neutrophil degranulation / proteolysis / extracellular space / extracellular region類似検索 - 分子機能 Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Papain-like cysteine endopeptidase / Cysteine peptidase, asparagine active site / Eukaryotic thiol (cysteine) proteases asparagine active site. / Cysteine peptidase, histidine active site / Eukaryotic thiol (cysteine) proteases histidine active site. / : / Peptidase C1A, papain C-terminal ...Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Papain-like cysteine endopeptidase / Cysteine peptidase, asparagine active site / Eukaryotic thiol (cysteine) proteases asparagine active site. / Cysteine peptidase, histidine active site / Eukaryotic thiol (cysteine) proteases histidine active site. / : / Peptidase C1A, papain C-terminal / Papain family cysteine protease / Papain family cysteine protease / Cysteine peptidase, cysteine active site / Eukaryotic thiol (cysteine) proteases cysteine active site. / Papain-like cysteine peptidase superfamily類似検索 - ドメイン・相同性 ACETATE ION / MALONIC ACID / Chem-Q1H / SUCCINIC ACID / Cathepsin S類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  フーリエ合成 / 解像度: 2.05 Å フーリエ合成 / 解像度: 2.05 Å |
|---|
 データ登録者 データ登録者 | Wagener, M. / Schade, M. / Merla, B. / Hars, U. / Kueckelhaus, S.Q. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2020 ジャーナル: J.Med.Chem. / 年: 2020
タイトル: Highly Selective Sub-Nanomolar Cathepsin S Inhibitors by Merging Fragment Binders with Nitrile Inhibitors.
著者: Schade, M. / Merla, B. / Lesch, B. / Wagener, M. / Timmermanns, S. / Pletinckx, K. / Hertrampf, T. |
|---|
| 履歴 | | 登録 | 2020年5月5日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2021年5月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年11月23日 | Group: Database references / Derived calculations
カテゴリ: atom_type / citation ...atom_type / citation / citation_author / database_2
Item: _atom_type.pdbx_N_electrons / _atom_type.pdbx_scat_Z ..._atom_type.pdbx_N_electrons / _atom_type.pdbx_scat_Z / _citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 1.2 | 2024年5月1日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
| 改定 1.3 | 2024年10月16日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 2.05 Å
フーリエ合成 / 解像度: 2.05 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2020
ジャーナル: J.Med.Chem. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6yyp.cif.gz
6yyp.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6yyp.ent.gz
pdb6yyp.ent.gz PDB形式
PDB形式 6yyp.json.gz
6yyp.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6yyp_validation.pdf.gz
6yyp_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6yyp_full_validation.pdf.gz
6yyp_full_validation.pdf.gz 6yyp_validation.xml.gz
6yyp_validation.xml.gz 6yyp_validation.cif.gz
6yyp_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/yy/6yyp
https://data.pdbj.org/pub/pdb/validation_reports/yy/6yyp ftp://data.pdbj.org/pub/pdb/validation_reports/yy/6yyp
ftp://data.pdbj.org/pub/pdb/validation_reports/yy/6yyp リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: CTSS / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CTSS / 発現宿主: 








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID29 / 波長: 0.97625 Å
/ ビームライン: ID29 / 波長: 0.97625 Å 解析
解析 フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj
















