+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6yvt | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | HIF prolyl hydroxylase 2 (PHD2/ EGLN1) in complex with MD-253 | ||||||
 要素 要素 | Egl nine homolog 1 | ||||||
 キーワード キーワード | OXIDOREDUCTASE / NON-HEME DIOXYGENASE / IRON / 2-OXOGLUTARATE / HYPOXIA-INDUCIBLE FACTOR / HIF / HIF PROLYL HYDROXYLASE DOMAIN 2 / PHD2 / EGLN1 / OXYGENASE / HYPOXIA / DNA-BINDING / METAL-BINDING / TRANSCRIPTION / HELIX-LOOP-HELIX-BETA / DSBH / FACIAL TRIAD / CYTOPLASM / TRANSCRIPTION/EPIGENETIC REGULATION / SIGNALING / DEVELOPMENT / CELL STRUCTURE / BETA-HYDROXYLATION / TRANSCRIPTION ACTIVATOR/INHIBITOR / UBL CONJUGATION / POLYMORPHISM / VITAMIN C / ZINC-FINGER / FAMILIAL ERYTHROCYTOSIS / BREAST CANCER / TRANSCRIPTION COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peptidyl-proline 4-dioxygenase activity / hypoxia-inducible factor-proline dioxygenase activity / hypoxia-inducible factor-proline dioxygenase / negative regulation of hypoxia-inducible factor-1alpha signaling pathway / peptidyl-proline dioxygenase activity / regulation protein catabolic process at postsynapse / intracellular oxygen homeostasis / labyrinthine layer development / regulation of modification of postsynaptic structure / heart trabecula formation ...peptidyl-proline 4-dioxygenase activity / hypoxia-inducible factor-proline dioxygenase activity / hypoxia-inducible factor-proline dioxygenase / negative regulation of hypoxia-inducible factor-1alpha signaling pathway / peptidyl-proline dioxygenase activity / regulation protein catabolic process at postsynapse / intracellular oxygen homeostasis / labyrinthine layer development / regulation of modification of postsynaptic structure / heart trabecula formation / 2-oxoglutarate-dependent dioxygenase activity / cardiac muscle tissue morphogenesis / L-ascorbic acid binding / response to nitric oxide / ventricular septum morphogenesis / regulation of neuron apoptotic process / enzyme inhibitor activity / regulation of angiogenesis / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / ferrous iron binding / cellular response to hypoxia / intracellular iron ion homeostasis / response to hypoxia / postsynaptic density / intracellular membrane-bounded organelle / glutamatergic synapse / enzyme binding / positive regulation of transcription by RNA polymerase II / zinc ion binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.847 Å 分子置換 / 解像度: 2.847 Å | ||||||
 データ登録者 データ登録者 | Chowdhury, R. / Demetriades, M. / Schofield, C.J. | ||||||
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2012 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2012タイトル: Dynamic combinatorial chemistry employing boronic acids/boronate esters leads to potent oxygenase inhibitors. 著者: Demetriades, M. / Leung, I.K. / Chowdhury, R. / Chan, M.C. / McDonough, M.A. / Yeoh, K.K. / Tian, Y.M. / Claridge, T.D. / Ratcliffe, P.J. / Woon, E.C. / Schofield, C.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6yvt.cif.gz 6yvt.cif.gz | 435.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6yvt.ent.gz pdb6yvt.ent.gz | 359.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6yvt.json.gz 6yvt.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6yvt_validation.pdf.gz 6yvt_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6yvt_full_validation.pdf.gz 6yvt_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  6yvt_validation.xml.gz 6yvt_validation.xml.gz | 47.1 KB | 表示 | |
| CIF形式データ |  6yvt_validation.cif.gz 6yvt_validation.cif.gz | 60.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yv/6yvt https://data.pdbj.org/pub/pdb/validation_reports/yv/6yvt ftp://data.pdbj.org/pub/pdb/validation_reports/yv/6yvt ftp://data.pdbj.org/pub/pdb/validation_reports/yv/6yvt | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2g19S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 6分子 ABCDEF
| #1: タンパク質 | 分子量: 28096.941 Da / 分子数: 6 / 由来タイプ: 組換発現 / 詳細: CATALYTIC DOMAIN (RESIDUES 181-426) / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: EGLN1, C1orf12, PNAS-118, PNAS-137 / プラスミド: PET28A(+) / 発現宿主: Homo sapiens (ヒト) / 遺伝子: EGLN1, C1orf12, PNAS-118, PNAS-137 / プラスミド: PET28A(+) / 発現宿主:  参照: UniProt: Q9GZT9, hypoxia-inducible factor-proline dioxygenase |
|---|
-非ポリマー , 5種, 71分子 
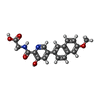







| #2: 化合物 | ChemComp-MN / #3: 化合物 | ChemComp-PW2 / #4: 化合物 | ChemComp-GOL / | #5: 化合物 | ChemComp-SO4 / | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.2 Å3/Da / 溶媒含有率: 61.53 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 6.5 詳細: Sample: 19.0 mg/ml PHD2 (50 mM Tris.HCl pH 7.5), 1.0 mM MnCl2 and 1.2 mM compound. Reservoir: 1.88 M ammonium sulfate, 0.1 M MES pH 6.5 and 5-7% dioxoane (v/v). Cryo-protection: 30% v/v glycerol. |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: RIGAKU SATURN 944 / 検出器: CCD / 日付: 2011年5月6日 / 詳細: VARIMAX HF | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: CONFOCAL / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.847→50 Å / Num. obs: 40604 / % possible obs: 99.4 % / 冗長度: 5.3 % / Biso Wilson estimate: 48.4 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.18 / Rpim(I) all: 0.112 / Rrim(I) all: 0.2 / Χ2: 1.098 / Net I/σ(I): 6.71 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 2G19 解像度: 2.847→23.753 Å / SU ML: 0.44 / 交差検証法: THROUGHOUT / σ(F): 1.96 / 位相誤差: 27.14 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT MODEL / Bsol: 57.9 Å2 / ksol: 0.45 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 211.06 Å2 / Biso mean: 55.0728 Å2 / Biso min: 19.96 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.847→23.753 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj




