| 登録情報 | データベース: PDB / ID: 6yso
|
|---|
| タイトル | Crystal structure of the (SR) Ca2+-ATPase solved by vanadium SAD phasing |
|---|
 要素 要素 | Sarcoplasmic/endoplasmic reticulum calcium ATPase 1 |
|---|
 キーワード キーワード | HYDROLASE / P-type ATPase / Vanadate / SAD phasing |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of cardiac muscle cell contraction / positive regulation of calcium ion import into sarcoplasmic reticulum / positive regulation of ATPase-coupled calcium transmembrane transporter activity / positive regulation of fast-twitch skeletal muscle fiber contraction / H zone / regulation of striated muscle contraction / calcium ion import into sarcoplasmic reticulum / negative regulation of striated muscle contraction / P-type Ca2+ transporter / P-type calcium transporter activity ...positive regulation of cardiac muscle cell contraction / positive regulation of calcium ion import into sarcoplasmic reticulum / positive regulation of ATPase-coupled calcium transmembrane transporter activity / positive regulation of fast-twitch skeletal muscle fiber contraction / H zone / regulation of striated muscle contraction / calcium ion import into sarcoplasmic reticulum / negative regulation of striated muscle contraction / P-type Ca2+ transporter / P-type calcium transporter activity / I band / endoplasmic reticulum-Golgi intermediate compartment / sarcoplasmic reticulum membrane / sarcoplasmic reticulum / calcium ion transport / calcium ion binding / endoplasmic reticulum membrane / perinuclear region of cytoplasm / endoplasmic reticulum / ATP hydrolysis activity / ATP binding / membrane類似検索 - 分子機能 P-type ATPase, subfamily IIA, SERCA-type / haloacid dehalogenase-like hydrolase / Calcium-transporting ATPase, cytoplasmic domain N / Calcium-transporting ATPase, cytoplasmic domain N / Cation-transporting P-type ATPase, C-terminal / Cation transporting ATPase, C-terminus / Cation transporter/ATPase, N-terminus / Cation-transporting P-type ATPase, N-terminal / Cation transporter/ATPase, N-terminus / Cation transport ATPase (P-type) ...P-type ATPase, subfamily IIA, SERCA-type / haloacid dehalogenase-like hydrolase / Calcium-transporting ATPase, cytoplasmic domain N / Calcium-transporting ATPase, cytoplasmic domain N / Cation-transporting P-type ATPase, C-terminal / Cation transporting ATPase, C-terminus / Cation transporter/ATPase, N-terminus / Cation-transporting P-type ATPase, N-terminal / Cation transporter/ATPase, N-terminus / Cation transport ATPase (P-type) / E1-E2 ATPase / P-type ATPase, haloacid dehalogenase domain / P-type ATPase, phosphorylation site / P-type ATPase, cytoplasmic domain N / E1-E2 ATPases phosphorylation site. / P-type ATPase, A domain superfamily / P-type ATPase / P-type ATPase, transmembrane domain superfamily / haloacid dehalogenase-like hydrolase / HAD superfamily / HAD-like superfamily / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Chem-128 / : / Chem-TG1 / oxido(dioxo)vanadium / Sarcoplasmic/endoplasmic reticulum calcium ATPase 1類似検索 - 構成要素 |
|---|
| 生物種 |   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 3.13 Å 単波長異常分散 / 解像度: 3.13 Å |
|---|
 データ登録者 データ登録者 | El Omari, K. / Mohamad, N. / Bountra, K. / Duman, R. / Romano, M. / Schlegel, K. / Kwong, H. / Mykhaylyk, V. / Olesen, C.E. / Moller, J.V. ...El Omari, K. / Mohamad, N. / Bountra, K. / Duman, R. / Romano, M. / Schlegel, K. / Kwong, H. / Mykhaylyk, V. / Olesen, C.E. / Moller, J.V. / Bublitz, M. / Beis, K. / Wagner, A. |
|---|
 引用 引用 |  ジャーナル: Iucrj / 年: 2020 ジャーナル: Iucrj / 年: 2020
タイトル: Experimental phasing with vanadium and application to nucleotide-binding membrane proteins.
著者: El Omari, K. / Mohamad, N. / Bountra, K. / Duman, R. / Romano, M. / Schlegel, K. / Kwong, H.S. / Mykhaylyk, V. / Olesen, C. / Moller, J.V. / Bublitz, M. / Beis, K. / Wagner, A. |
|---|
| 履歴 | | 登録 | 2020年4月22日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2020年11月4日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年12月2日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.name |
|---|
| 改定 1.2 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 3.13 Å
単波長異常分散 / 解像度: 3.13 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Iucrj / 年: 2020
ジャーナル: Iucrj / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6yso.cif.gz
6yso.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6yso.ent.gz
pdb6yso.ent.gz PDB形式
PDB形式 6yso.json.gz
6yso.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6yso_validation.pdf.gz
6yso_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6yso_full_validation.pdf.gz
6yso_full_validation.pdf.gz 6yso_validation.xml.gz
6yso_validation.xml.gz 6yso_validation.cif.gz
6yso_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ys/6yso
https://data.pdbj.org/pub/pdb/validation_reports/ys/6yso ftp://data.pdbj.org/pub/pdb/validation_reports/ys/6yso
ftp://data.pdbj.org/pub/pdb/validation_reports/ys/6yso リンク
リンク 集合体
集合体


 要素
要素

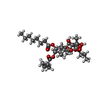










 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I23 / 波長: 2.26009 Å
/ ビームライン: I23 / 波長: 2.26009 Å 解析
解析 単波長異常分散 / 解像度: 3.13→64.585 Å / SU ML: 0.51 / 交差検証法: THROUGHOUT / σ(F): 1.33 / 位相誤差: 29.6
単波長異常分散 / 解像度: 3.13→64.585 Å / SU ML: 0.51 / 交差検証法: THROUGHOUT / σ(F): 1.33 / 位相誤差: 29.6  ムービー
ムービー コントローラー
コントローラー
























 PDBj
PDBj







