+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6yie | ||||||
|---|---|---|---|---|---|---|---|
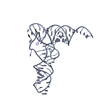
| タイトル | Structure of a Borealin-INCENP-Survivin complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | CELL CYCLE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報central element / meiotic spindle midzone / survivin complex / meiotic spindle midzone assembly / : / positive regulation of mitotic sister chromatid separation / positive regulation of mitotic cytokinesis / metaphase chromosome alignment / positive regulation of mitotic cell cycle spindle assembly checkpoint / positive regulation of exit from mitosis ...central element / meiotic spindle midzone / survivin complex / meiotic spindle midzone assembly / : / positive regulation of mitotic sister chromatid separation / positive regulation of mitotic cytokinesis / metaphase chromosome alignment / positive regulation of mitotic cell cycle spindle assembly checkpoint / positive regulation of exit from mitosis / mitotic spindle midzone assembly / positive regulation of attachment of mitotic spindle microtubules to kinetochore / chromocenter / interphase microtubule organizing center / lateral element / chromosome passenger complex / protein-containing complex localization / cysteine-type endopeptidase inhibitor activity involved in apoptotic process / cobalt ion binding / mitotic metaphase chromosome alignment / nuclear chromosome / mitotic spindle assembly checkpoint signaling / spindle midzone / pericentric heterochromatin / mitotic cytokinesis / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / mitotic sister chromatid segregation / SUMOylation of DNA replication proteins / chromosome, centromeric region / cysteine-type endopeptidase inhibitor activity / mitotic spindle assembly / chromosome organization / intercellular bridge / cytoplasmic microtubule / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / centriole / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / tubulin binding / positive regulation of mitotic cell cycle / Resolution of Sister Chromatid Cohesion / molecular function activator activity / mitotic spindle organization / spindle microtubule / chromosome segregation / RHO GTPases Activate Formins / sensory perception of sound / kinetochore / small GTPase binding / spindle / G2/M transition of mitotic cell cycle / Separation of Sister Chromatids / Neddylation / mitotic cell cycle / protein-folding chaperone binding / microtubule cytoskeleton / midbody / Interleukin-4 and Interleukin-13 signaling / microtubule binding / microtubule / nuclear body / protein heterodimerization activity / cell division / negative regulation of DNA-templated transcription / positive regulation of cell population proliferation / apoptotic process / negative regulation of apoptotic process / nucleolus / enzyme binding / protein homodimerization activity / protein-containing complex / zinc ion binding / nucleoplasm / metal ion binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.49 Å 分子置換 / 解像度: 3.49 Å | ||||||
 データ登録者 データ登録者 | Serena, M. / Elliott, P.R. / Barr, F.A. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: J.Cell Biol. / 年: 2020 ジャーナル: J.Cell Biol. / 年: 2020タイトル: Molecular basis of MKLP2-dependent Aurora B transport from chromatin to the anaphase central spindle. 著者: Serena, M. / Bastos, R.N. / Elliott, P.R. / Barr, F.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6yie.cif.gz 6yie.cif.gz | 318.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6yie.ent.gz pdb6yie.ent.gz | 219.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6yie.json.gz 6yie.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6yie_validation.pdf.gz 6yie_validation.pdf.gz | 470.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6yie_full_validation.pdf.gz 6yie_full_validation.pdf.gz | 474.4 KB | 表示 | |
| XML形式データ |  6yie_validation.xml.gz 6yie_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  6yie_validation.cif.gz 6yie_validation.cif.gz | 23.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yi/6yie https://data.pdbj.org/pub/pdb/validation_reports/yi/6yie ftp://data.pdbj.org/pub/pdb/validation_reports/yi/6yie ftp://data.pdbj.org/pub/pdb/validation_reports/yi/6yie | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCSアンサンブル:
|
- 要素
要素
| #1: タンパク質 | 分子量: 16568.902 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: BIRC5, API4, IAP4 / プラスミド: pETDuet1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: BIRC5, API4, IAP4 / プラスミド: pETDuet1 / 発現宿主:  #2: タンパク質 | 分子量: 11617.326 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CDCA8, PESCRG3 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CDCA8, PESCRG3 / 発現宿主:  #3: タンパク質 | 分子量: 7018.110 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: INCENP / プラスミド: PFAT2-His6-GST / 発現宿主: Homo sapiens (ヒト) / 遺伝子: INCENP / プラスミド: PFAT2-His6-GST / 発現宿主:  #4: 化合物 | 研究の焦点であるリガンドがあるか | N | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.09 Å3/Da / 溶媒含有率: 41.11 % |
|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法 / pH: 6 / 詳細: 5 % (w/v) PEG 3,350, 50 mM MES pH 6.0 |
-データ収集
| 回折 | 平均測定温度: 110 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / ビームライン: P14 (MX2) / 波長: 0.9763 Å / ビームライン: P14 (MX2) / 波長: 0.9763 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2017年6月21日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9763 Å / 相対比: 1 |
| 反射 | 解像度: 3.49→78.82 Å / Num. obs: 6947 / % possible obs: 88.5 % / 冗長度: 3.7 % / Biso Wilson estimate: 65.27 Å2 / CC1/2: 0.992 / Rmerge(I) obs: 0.162 / Net I/σ(I): 3.5 |
| 反射 シェル | 解像度: 3.49→3.83 Å / Rmerge(I) obs: 0.435 / Mean I/σ(I) obs: 1.7 / Num. unique obs: 1644 / CC1/2: 0.902 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 2QFA 解像度: 3.49→66.7 Å / SU ML: 0.4279 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 35.3698
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 64.38 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.49→66.7 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj














