| #1: タンパク質 | Erythromycin resistance repressor protein / Macrolide 2'-phosphotransferase / Macrolide 2'-phosphotransferase MphR(A) / MphR(A) / MphR(A) ...Macrolide 2'-phosphotransferase / Macrolide 2'-phosphotransferase MphR(A) / MphR(A) / MphR(A) erythromycin resistance repressor protein / Regulator of macrolide 2'-phosphotransferase I / Repressor protein MphR(A) / TetR/AcrR family transcriptional regulator / Transcriptional regulator / TetR family
分子量: 21601.975 Da / 分子数: 2 / 変異: R122T, K132N, A151T, H184Q / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌)遺伝子: mphR(A), mphR, A9R57_28010, ACN77_02555, ACN77_03810, ACN77_19745, AKG29_00740, AM465_27045, APT94_14580, B1K96_22845, B9M99_28120, B9N33_26285, B9T59_28650, BANRA_01808, BANRA_05330, ...遺伝子: mphR(A), mphR, A9R57_28010, ACN77_02555, ACN77_03810, ACN77_19745, AKG29_00740, AM465_27045, APT94_14580, B1K96_22845, B9M99_28120, B9N33_26285, B9T59_28650, BANRA_01808, BANRA_05330, BANRA_05395, BE963_00530, BEN53_26070, BET08_23460, BFD68_26430, BJJ90_27670, BK248_28255, BK292_28800, BK334_26705, BK373_25645, BK375_28450, BK383_16415, BK400_25090, BVL39_07785, BVL39_26565, C5N07_27930, C5P01_27745, C5P43_14800, C5P44_28315, C6669_28285, C6986_31345, C7235_25300, C7235_26770, CQP61_00550, CR538_26710, CRT43_27530, CRT46_27035, CWS33_26295, D0X26_00055, D0X26_28400, D3O91_28140, D9N32_01235, DB359_27590, DBY14_07355, DBY14_07475, DBY14_07595, DBY14_07715, DBY14_07835, DBY14_07955, DBY14_08075, DBY14_08195, DBY14_08400, DBY14_09000, DBY14_09120, DBY14_09240, DBY14_09360, DBY14_09480, DBY14_09600, DBY14_09720, DBY14_09840, DBY14_09960, DBY14_10080, DBY14_10200, DBY14_10320, DBY14_10440, DBY14_10560, DBY14_10680, DBY14_10800, DBY14_10920, DBY14_11040, DBY14_11160, DBY14_11280, DL545_01125, DL545_26270, DM102_26995, DM129_26600, DM129_28140, DNQ41_27340, DS732_30515, E4Z89_24290, Eco118UI_26585, ECONIH1_26780, ECTO124_05297, EGT48_01470, EIA21_26670, EJC48_00900, EJC75_00560, EJH97_25820, EPS71_24560, EQ823_24865, EQ830_27025, ERS085366_04056, ERS085404_05054, ERS150873_04751, ETN48_p0081, EVY21_24885, ExPECSC022_02556, ExPECSC036_03428, ExPECSC065_05093, EXX13_27025, EYD11_23970, FORC82_p029, HmCmsJML146_04799, HMPREF3040_01640, MS6198_B124, MS8345_A00020, NCTC13846_04922, U12A_A00163, U14A_A00115, UN86_19865, YDC107_4984発現宿主 Escherichia coli (大腸菌)遺伝子: mphR(A), mphR, A9R57_28010, ACN77_02555, ACN77_03810, ACN77_19745, AKG29_00740, AM465_27045, APT94_14580, B1K96_22845, B9M99_28120, B9N33_26285, B9T59_28650, BANRA_01808, BANRA_05330, ...遺伝子: mphR(A), mphR, A9R57_28010, ACN77_02555, ACN77_03810, ACN77_19745, AKG29_00740, AM465_27045, APT94_14580, B1K96_22845, B9M99_28120, B9N33_26285, B9T59_28650, BANRA_01808, BANRA_05330, BANRA_05395, BE963_00530, BEN53_26070, BET08_23460, BFD68_26430, BJJ90_27670, BK248_28255, BK292_28800, BK334_26705, BK373_25645, BK375_28450, BK383_16415, BK400_25090, BVL39_07785, BVL39_26565, C5N07_27930, C5P01_27745, C5P43_14800, C5P44_28315, C6669_28285, C6986_31345, C7235_25300, C7235_26770, CQP61_00550, CR538_26710, CRT43_27530, CRT46_27035, CWS33_26295, D0X26_00055, D0X26_28400, D3O91_28140, D9N32_01235, DB359_27590, DBY14_07355, DBY14_07475, DBY14_07595, DBY14_07715, DBY14_07835, DBY14_07955, DBY14_08075, DBY14_08195, DBY14_08400, DBY14_09000, DBY14_09120, DBY14_09240, DBY14_09360, DBY14_09480, DBY14_09600, DBY14_09720, DBY14_09840, DBY14_09960, DBY14_10080, DBY14_10200, DBY14_10320, DBY14_10440, DBY14_10560, DBY14_10680, DBY14_10800, DBY14_10920, DBY14_11040, DBY14_11160, DBY14_11280, DL545_01125, DL545_26270, DM102_26995, DM129_26600, DM129_28140, DNQ41_27340, DS732_30515, E4Z89_24290, Eco118UI_26585, ECONIH1_26780, ECTO124_05297, EGT48_01470, EIA21_26670, EJC48_00900, EJC75_00560, EJH97_25820, EPS71_24560, EQ823_24865, EQ830_27025, ERS085366_04056, ERS085404_05054, ERS150873_04751, ETN48_p0081, EVY21_24885, ExPECSC022_02556, ExPECSC036_03428, ExPECSC065_05093, EXX13_27025, EYD11_23970, FORC82_p029, HmCmsJML146_04799, HMPREF3040_01640, MS6198_B124, MS8345_A00020, NCTC13846_04922, U12A_A00163, U14A_A00115, UN86_19865, YDC107_4984発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q9EVJ6       | #2: 化合物 | ChemComp-CTY / CLARITHROMYCIN / クラリスロマイシン
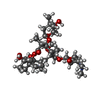  分子量 分子量: 747.953 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C 38H 69NO 13 / タイプ: SUBJECT OF INVESTIGATION / コメント: 抗生剤 *YM| #3: 化合物 | ChemComp-FLC / CITRATE ANION / シトラ-ト
  分子量 分子量: 189.100 Da / 分子数: 1 / 由来タイプ: 天然 / 式: C 6H 5O 7 |
|---|
| #4: 水 | ChemComp-HOH / water
 分子量 分子量: 18.015 Da / 分子数: 135 / 由来タイプ: 天然 / 式: H 2O |
|---|
| 研究の焦点であるリガンドがあるか | Y |
|---|
|
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Acs Synth Biol / 年: 2021
ジャーナル: Acs Synth Biol / 年: 2021 ジャーナル: J.Mol.Biol. / 年: 2009
ジャーナル: J.Mol.Biol. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6u18.cif.gz
6u18.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6u18.ent.gz
pdb6u18.ent.gz PDB形式
PDB形式 6u18.json.gz
6u18.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6u18_validation.pdf.gz
6u18_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6u18_full_validation.pdf.gz
6u18_full_validation.pdf.gz 6u18_validation.xml.gz
6u18_validation.xml.gz 6u18_validation.cif.gz
6u18_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/u1/6u18
https://data.pdbj.org/pub/pdb/validation_reports/u1/6u18 ftp://data.pdbj.org/pub/pdb/validation_reports/u1/6u18
ftp://data.pdbj.org/pub/pdb/validation_reports/u1/6u18
 リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5419 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5419 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj










