+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6rwx | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
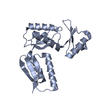
| タイトル | Periplasmic inner membrane ring of the Shigella type 3 secretion system | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | PROTEIN TRANSPORT / type 3 secretion system / shigella / ring-forming membrane protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.55 Å | |||||||||
 データ登録者 データ登録者 | Kamprad, A. / Lunelli, M. | |||||||||
| 資金援助 | 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2020 ジャーナル: PLoS Pathog / 年: 2020タイトル: Cryo-EM structure of the Shigella type III needle complex. 著者: Michele Lunelli / Antje Kamprad / Jörg Bürger / Thorsten Mielke / Christian M T Spahn / Michael Kolbe /  要旨: The Type III Secretion Systems (T3SS) needle complex is a conserved syringe-shaped protein translocation nanomachine with a mass of about 3.5 MDa essential for the survival and virulence of many Gram- ...The Type III Secretion Systems (T3SS) needle complex is a conserved syringe-shaped protein translocation nanomachine with a mass of about 3.5 MDa essential for the survival and virulence of many Gram-negative bacterial pathogens. This system is composed of a membrane-embedded basal body and an extracellular needle that deliver effector proteins into host cells. High-resolution structures of the T3SS from different organisms and infection stages are needed to understand the underlying molecular mechanisms of effector translocation. Here, we present the cryo-electron microscopy structure of the isolated Shigella T3SS needle complex. The inner membrane (IM) region of the basal body adopts 24-fold rotational symmetry and forms a channel system that connects the bacterial periplasm with the export apparatus cage. The secretin oligomer adopts a heterogeneous architecture with 16- and 15-fold cyclic symmetry in the periplasmic N-terminal connector and C-terminal outer membrane ring, respectively. Two out of three IM subunits bind the secretin connector via a β-sheet augmentation. The cryo-EM map also reveals the helical architecture of the export apparatus core, the inner rod, the needle and their intervening interfaces. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6rwx.cif.gz 6rwx.cif.gz | 1.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6rwx.ent.gz pdb6rwx.ent.gz | 1.2 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6rwx.json.gz 6rwx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6rwx_validation.pdf.gz 6rwx_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6rwx_full_validation.pdf.gz 6rwx_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  6rwx_validation.xml.gz 6rwx_validation.xml.gz | 204.4 KB | 表示 | |
| CIF形式データ |  6rwx_validation.cif.gz 6rwx_validation.cif.gz | 275.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rw/6rwx https://data.pdbj.org/pub/pdb/validation_reports/rw/6rwx ftp://data.pdbj.org/pub/pdb/validation_reports/rw/6rwx ftp://data.pdbj.org/pub/pdb/validation_reports/rw/6rwx | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 43053.844 Da / 分子数: 24 / 由来タイプ: 天然 由来: (天然)  Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌)Plasmid details: encoded on pWR100 plasmid / Variant: M90T / 参照: UniProt: P0A221 #2: タンパク質 | 分子量: 27542.055 Da / 分子数: 24 / 由来タイプ: 天然 由来: (天然)  Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌)Variant: M90T / 参照: UniProt: Q06081 Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: The periplasmic inner membrane ring of the Shigella type 3 secretion system タイプ: COMPLEX 詳細: Focused reconstruction from isolated needle complexes of the Shigella type 3 secretion system Entity ID: all / 由来: NATURAL |
|---|---|
| 分子量 | 単位: MEGADALTONS / 実験値: NO |
| 由来(天然) | 生物種:  Shigella flexneri (フレクスナー赤痢菌) / 株: M90T / 細胞内の位置: Membrane Shigella flexneri (フレクスナー赤痢菌) / 株: M90T / 細胞内の位置: Membrane |
| 緩衝液 | pH: 8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES / 詳細: Isolated needle complex in detergent solution |
| 試料支持 | グリッドの材料: COPPER / グリッドのタイプ: Quantifoil R2/1 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 295 K 詳細: Sample applied on grid 5 ul, incubation time 5 min on ice, then moved into Vitrobot and 5 ul sample applied again. Blot time: 2 sec Blot force: -2 Drain time: 0 sec |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 101179 X / 最大 デフォーカス(公称値): 4 nm / 最小 デフォーカス(公称値): 1.5 nm / Cs: 2.7 mm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 1.5 sec. / 電子線照射量: 25 e/Å2 フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 撮影したグリッド数: 1 / 実像数: 5238 |
| 画像スキャン | 横: 4096 / 縦: 4096 / 動画フレーム数/画像: 7 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.13_2998: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 171833 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C24 (24回回転対称) | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.55 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 72298 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 140 / プロトコル: AB INITIO MODEL / 空間: REAL / Target criteria: Correlation coefficient and geometry 詳細: Model was built and refined in the map low-pass filtered at 3.2 A | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1 / Source name: PDB / タイプ: experimental model
| ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj