| 登録情報 | データベース: PDB / ID: 6qtm
|
|---|
| タイトル | Crystal structure of the Sir4 H-BRCT domain in complex with Ty5 pS1095 peptide |
|---|
 要素 要素 | - Regulatory protein SIR4
- Ribonuclease H
|
|---|
 キーワード キーワード | NUCLEAR PROTEIN / Heterochromatin |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
transposition / establishment of protein-containing complex localization to telomere / telomere tethering at nuclear periphery / chromatin silencing complex / silent mating-type cassette heterochromatin formation / positive regulation of heterochromatin formation / subtelomeric heterochromatin formation / heterochromatin formation / nucleosome binding / DNA integration ...transposition / establishment of protein-containing complex localization to telomere / telomere tethering at nuclear periphery / chromatin silencing complex / silent mating-type cassette heterochromatin formation / positive regulation of heterochromatin formation / subtelomeric heterochromatin formation / heterochromatin formation / nucleosome binding / DNA integration / double-strand break repair via nonhomologous end joining / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / double-stranded DNA binding / chromosome, telomeric region / molecular adaptor activity / DNA-directed DNA polymerase activity / RNA binding / nucleus / cytoplasm類似検索 - 分子機能 GAG-pre-integrase domain / Protein of unknown function DUF5314 / GAG-pre-integrase domain / Ty5 Gag N-terminal region / Sir4, SID domain / Sir4 SID domain / Reverse transcriptase, RNA-dependent DNA polymerase / Reverse transcriptase (RNA-dependent DNA polymerase) / Integrase core domain / Integrase, catalytic core ...GAG-pre-integrase domain / Protein of unknown function DUF5314 / GAG-pre-integrase domain / Ty5 Gag N-terminal region / Sir4, SID domain / Sir4 SID domain / Reverse transcriptase, RNA-dependent DNA polymerase / Reverse transcriptase (RNA-dependent DNA polymerase) / Integrase core domain / Integrase, catalytic core / Integrase catalytic domain profile. / Ribonuclease H superfamily / Ribonuclease H-like superfamily / DNA/RNA polymerase superfamily類似検索 - ドメイン・相同性 Integrase catalytic domain-containing protein / Regulatory protein SIR4類似検索 - 構成要素 |
|---|
| 生物種 |   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母)
  Saccharomyces paradoxus (酵母) Saccharomyces paradoxus (酵母) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3 Å 分子置換 / 解像度: 3 Å |
|---|
 データ登録者 データ登録者 | Gut, H. / Deshpande, I. / Keusch, J.J. / Challa, K. / Iesmantavicius, V. / Gasser, S.M. |
|---|
 引用 引用 |  ジャーナル: Embo J. / 年: 2019 ジャーナル: Embo J. / 年: 2019
タイトル: The Sir4 H-BRCT domain interacts with phospho-proteins to sequester and repress yeast heterochromatin.
著者: Deshpande, I. / Keusch, J.J. / Challa, K. / Iesmantavicius, V. / Gasser, S.M. / Gut, H. |
|---|
| 履歴 | | 登録 | 2019年2月25日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年9月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年9月25日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.page_first / _citation.page_last ..._citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2019年10月23日 | Group: Data collection / Database references / カテゴリ: citation / Item: _citation.journal_volume |
|---|
| 改定 1.3 | 2021年8月11日 | Group: Database references / Derived calculations ...Database references / Derived calculations / Source and taxonomy / Structure summary
カテゴリ: database_2 / entity ...database_2 / entity / entity_name_com / pdbx_entity_src_syn / pdbx_struct_mod_residue / struct_ref / struct_ref_seq
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _entity.pdbx_description / _entity.pdbx_ec / _pdbx_entity_src_syn.ncbi_taxonomy_id / _pdbx_entity_src_syn.organism_common_name / _pdbx_entity_src_syn.organism_scientific / _struct_ref.db_code / _struct_ref.db_name / _struct_ref.pdbx_align_begin / _struct_ref.pdbx_db_accession / _struct_ref.pdbx_seq_one_letter_code / _struct_ref_seq.pdbx_db_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3 Å
分子置換 / 解像度: 3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Embo J. / 年: 2019
ジャーナル: Embo J. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6qtm.cif.gz
6qtm.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6qtm.ent.gz
pdb6qtm.ent.gz PDB形式
PDB形式 6qtm.json.gz
6qtm.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6qtm_validation.pdf.gz
6qtm_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6qtm_full_validation.pdf.gz
6qtm_full_validation.pdf.gz 6qtm_validation.xml.gz
6qtm_validation.xml.gz 6qtm_validation.cif.gz
6qtm_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/qt/6qtm
https://data.pdbj.org/pub/pdb/validation_reports/qt/6qtm ftp://data.pdbj.org/pub/pdb/validation_reports/qt/6qtm
ftp://data.pdbj.org/pub/pdb/validation_reports/qt/6qtm リンク
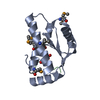
リンク 集合体
集合体
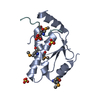


 要素
要素


 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06DA / 波長: 1 Å
/ ビームライン: X06DA / 波長: 1 Å 解析
解析 分子置換 / 解像度: 3→49.33 Å / Cor.coef. Fo:Fc: 0.8691 / Cor.coef. Fo:Fc free: 0.8367 / 交差検証法: THROUGHOUT / σ(F): 0 / SU Rfree Blow DPI: 0.43
分子置換 / 解像度: 3→49.33 Å / Cor.coef. Fo:Fc: 0.8691 / Cor.coef. Fo:Fc free: 0.8367 / 交差検証法: THROUGHOUT / σ(F): 0 / SU Rfree Blow DPI: 0.43  ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj



