[English] 日本語
 Yorodumi
Yorodumi- PDB-6qji: Crystal Structure of the third PDZ domain of PSD-95 protein: spac... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6qji | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
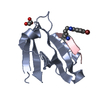
| Title | Crystal Structure of the third PDZ domain of PSD-95 protein: space group P3112 | |||||||||
 Components Components | Disks large homolog 4 | |||||||||
 Keywords Keywords | SIGNALING PROTEIN / pdz domain | |||||||||
| Function / homology |  Function and homology information Function and homology informationLGI-ADAM interactions / P2Y1 nucleotide receptor binding / beta-1 adrenergic receptor binding / neuroligin family protein binding / regulation of grooming behavior / NrCAM interactions / synaptic vesicle maturation / positive regulation of neuron projection arborization / receptor localization to synapse / vocalization behavior ...LGI-ADAM interactions / P2Y1 nucleotide receptor binding / beta-1 adrenergic receptor binding / neuroligin family protein binding / regulation of grooming behavior / NrCAM interactions / synaptic vesicle maturation / positive regulation of neuron projection arborization / receptor localization to synapse / vocalization behavior / neuron spine / Synaptic adhesion-like molecules / cerebellar mossy fiber / establishment or maintenance of epithelial cell apical/basal polarity / protein localization to synapse / Trafficking of AMPA receptors / dendritic spine morphogenesis / negative regulation of receptor internalization / juxtaparanode region of axon / acetylcholine receptor binding / RHO GTPases activate CIT / cellular response to potassium ion / Assembly and cell surface presentation of NMDA receptors / NMDA selective glutamate receptor signaling pathway / Neurexins and neuroligins / neuromuscular process controlling balance / Activation of Ca-permeable Kainate Receptor / neurotransmitter receptor localization to postsynaptic specialization membrane / cortical cytoskeleton / neuron projection terminus / Negative regulation of NMDA receptor-mediated neuronal transmission / Unblocking of NMDA receptors, glutamate binding and activation / AMPA glutamate receptor clustering / locomotory exploration behavior / Signaling by ERBB4 / AMPA glutamate receptor complex / Long-term potentiation / social behavior / D1 dopamine receptor binding / positive regulation of synaptic transmission / regulation of postsynaptic membrane neurotransmitter receptor levels / ionotropic glutamate receptor binding / excitatory synapse / dendrite cytoplasm / Ras activation upon Ca2+ influx through NMDA receptor / synaptic membrane / positive regulation of excitatory postsynaptic potential / learning / PDZ domain binding / adherens junction / neuromuscular junction / establishment of protein localization / cell-cell adhesion / regulation of long-term neuronal synaptic plasticity / postsynaptic density membrane / kinase binding / synaptic vesicle / endocytic vesicle membrane / cell junction / nervous system development / positive regulation of cytosolic calcium ion concentration / RAF/MAP kinase cascade / protein-containing complex assembly / scaffold protein binding / protein phosphatase binding / dendritic spine / chemical synaptic transmission / postsynaptic membrane / neuron projection / postsynaptic density / synapse / protein kinase binding / protein-containing complex binding / glutamatergic synapse / endoplasmic reticulum / signal transduction / plasma membrane / cytoplasm / cytosol Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.5 Å molecular replacement / Resolution: 1.5 Å | |||||||||
 Authors Authors | Camara-Artigas, A. | |||||||||
| Funding support |  Spain, 1items Spain, 1items
| |||||||||
 Citation Citation |  Journal: Acta Crystallogr D Struct Biol / Year: 2019 Journal: Acta Crystallogr D Struct Biol / Year: 2019Title: Conformational changes in the third PDZ domain of the neuronal postsynaptic density protein 95. Authors: Camara-Artigas, A. / Murciano-Calles, J. / Martinez, J.C. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6qji.cif.gz 6qji.cif.gz | 317.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6qji.ent.gz pdb6qji.ent.gz | 267.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6qji.json.gz 6qji.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/qj/6qji https://data.pdbj.org/pub/pdb/validation_reports/qj/6qji ftp://data.pdbj.org/pub/pdb/validation_reports/qj/6qji ftp://data.pdbj.org/pub/pdb/validation_reports/qj/6qji | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6qjdC  6qjfC  6qjgC  6qjjC  6qjkC  6qjlC  6qjnC  3k82S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||
| 2 | 
| |||||||||||||||
| 3 | 
| |||||||||||||||
| 4 | 
| |||||||||||||||
| 5 | 
| |||||||||||||||
| 6 | 
| |||||||||||||||
| Unit cell |
| |||||||||||||||
| Components on special symmetry positions |
|
- Components
Components
| #1: Protein | Mass: 10660.919 Da / Num. of mol.: 6 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: DLG4, PSD95 / Production host: Homo sapiens (human) / Gene: DLG4, PSD95 / Production host:  #2: Chemical | ChemComp-SO4 / #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.89 Å3/Da / Density % sol: 35 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 6 / Details: 0.2 M AMMONIUM SULPHATE, 30% PEG 4000 |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: ID14-4 / Wavelength: 0.9393 Å / Beamline: ID14-4 / Wavelength: 0.9393 Å | ||||||||||||||||||||||||||||||
| Detector | Type: ADSC QUANTUM 315r / Detector: CCD / Date: Apr 4, 2011 | ||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9393 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.5→19.97 Å / Num. obs: 80715 / % possible obs: 99.6 % / Redundancy: 7.7 % / CC1/2: 0.998 / Rmerge(I) obs: 0.072 / Rpim(I) all: 0.026 / Rrim(I) all: 0.077 / Net I/σ(I): 16.6 / Num. measured all: 618089 / Scaling rejects: 133 | ||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
-Phasing
| Phasing | Method:  molecular replacement molecular replacement |
|---|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3K82 Resolution: 1.5→19.972 Å / SU ML: 0.21 / Cross valid method: THROUGHOUT / σ(F): 0.08 / Phase error: 26.11 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 69.42 Å2 / Biso mean: 23.5923 Å2 / Biso min: 6.43 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.5→19.972 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 30
|
 Movie
Movie Controller
Controller











 PDBj
PDBj















