| Entry | Database: PDB / ID: 6pgl
|
|---|
| Title | Structure of Kluyveromyces marxianus Usb1 with uridine monophosphate |
|---|
 Components Components | Uncharacterized protein YLR132C |
|---|
 Keywords Keywords | HYDROLASE / exonuclease / U6 snRNA / 2H phosphodiesterase superfamily |
|---|
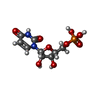
| Function / homology | U6 snRNA phosphodiesterase 1 / Uncharacterised conserved protein / U6 snRNA 3'-end processing / nuclease activity / Chem-PG6 / TRIETHYLENE GLYCOL / URIDINE-5'-MONOPHOSPHATE / Uncharacterized protein YLR132C Function and homology information Function and homology information |
|---|
| Biological species |  Kluyveromyces marxianus (yeast) Kluyveromyces marxianus (yeast) |
|---|
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.845 Å MOLECULAR REPLACEMENT / Resolution: 1.845 Å |
|---|
 Authors Authors | Nomura, Y. / Montemayor, E.J. / Butcher, S.E. |
|---|
| Funding support |  United States, 2items United States, 2items | Organization | Grant number | Country |
|---|
| National Institutes of Health/National Human Genome Research Institute (NIH/NHGRI) | R01 GM065166 |  United States United States | | National Institutes of Health/National Human Genome Research Institute (NIH/NHGRI) | R35 GM118131 |  United States United States |
|
|---|
 Citation Citation |  Journal: Nucleic Acids Res. / Year: 2020 Journal: Nucleic Acids Res. / Year: 2020
Title: Structural basis for the evolution of cyclic phosphodiesterase activity in the U6 snRNA exoribonuclease Usb1.
Authors: Nomura, Y. / Montemayor, E.J. / Virta, J.M. / Hayes, S.M. / Butcher, S.E. |
|---|
| History | | Deposition | Jun 24, 2019 | Deposition site: RCSB / Processing site: RCSB |
|---|
| Revision 1.0 | Dec 11, 2019 | Provider: repository / Type: Initial release |
|---|
| Revision 1.1 | May 20, 2020 | Group: Database references / Category: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| Revision 1.2 | Mar 13, 2024 | Group: Data collection / Database references / Category: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information Kluyveromyces marxianus (yeast)
Kluyveromyces marxianus (yeast) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.845 Å
MOLECULAR REPLACEMENT / Resolution: 1.845 Å  Authors
Authors United States, 2items
United States, 2items  Citation
Citation Journal: Nucleic Acids Res. / Year: 2020
Journal: Nucleic Acids Res. / Year: 2020 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 6pgl.cif.gz
6pgl.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb6pgl.ent.gz
pdb6pgl.ent.gz PDB format
PDB format 6pgl.json.gz
6pgl.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/pg/6pgl
https://data.pdbj.org/pub/pdb/validation_reports/pg/6pgl ftp://data.pdbj.org/pub/pdb/validation_reports/pg/6pgl
ftp://data.pdbj.org/pub/pdb/validation_reports/pg/6pgl Links
Links Assembly
Assembly
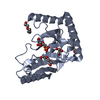


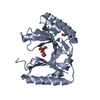
 Components
Components Kluyveromyces marxianus (strain DMKU3-1042 / BCC 29191 / NBRC 104275) (yeast)
Kluyveromyces marxianus (strain DMKU3-1042 / BCC 29191 / NBRC 104275) (yeast)











 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  APS
APS  / Beamline: 21-ID-F / Wavelength: 0.9787 Å
/ Beamline: 21-ID-F / Wavelength: 0.9787 Å Processing
Processing MOLECULAR REPLACEMENT / Resolution: 1.845→55.172 Å / SU ML: 0.19 / Cross valid method: FREE R-VALUE / σ(F): 1.9 / Phase error: 26.27
MOLECULAR REPLACEMENT / Resolution: 1.845→55.172 Å / SU ML: 0.19 / Cross valid method: FREE R-VALUE / σ(F): 1.9 / Phase error: 26.27  Movie
Movie Controller
Controller












 PDBj
PDBj







