+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6ng2 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of human neuronal nitric oxide synthase R354A/G357D mutant heme domain in complex with 6-(3-(3-(dimethylamino)prop-1-yn-1-yl)-5-fluorophenethyl)-4-methylpyridin-2-amine | ||||||
 要素 要素 | Nitric oxide synthase, brain | ||||||
 キーワード キーワード | OXIDOREDUCTASE/OXIDOREDUCTASE INHIBITOR / nitric oxide synthase / inhibitor complex / heme enzyme / OXIDOREDUCTASE-OXIDOREDUCTASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of membrane repolarization during ventricular cardiac muscle cell action potential / negative regulation of calcium ion transport into cytosol / Nitric oxide stimulates guanylate cyclase / myoblast fusion / ROS and RNS production in phagocytes / tetrahydrobiopterin binding / arginine binding / regulation of calcium ion transmembrane transport via high voltage-gated calcium channel / regulation of cardiac muscle contraction by calcium ion signaling / synaptic signaling by nitric oxide ...positive regulation of membrane repolarization during ventricular cardiac muscle cell action potential / negative regulation of calcium ion transport into cytosol / Nitric oxide stimulates guanylate cyclase / myoblast fusion / ROS and RNS production in phagocytes / tetrahydrobiopterin binding / arginine binding / regulation of calcium ion transmembrane transport via high voltage-gated calcium channel / regulation of cardiac muscle contraction by calcium ion signaling / synaptic signaling by nitric oxide / positive regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / positive regulation of sodium ion transmembrane transport / peptidyl-cysteine S-nitrosylase activity / cadmium ion binding / negative regulation of calcium ion transport / positive regulation of the force of heart contraction / negative regulation of potassium ion transport / regulation of postsynaptic membrane potential / nitric oxide mediated signal transduction / nitric-oxide synthase (NADPH) / sodium channel regulator activity / negative regulation of serotonin uptake / regulation of neurogenesis / regulation of cardiac muscle contraction / nitric-oxide synthase activity / multicellular organismal response to stress / xenobiotic catabolic process / L-arginine catabolic process / striated muscle contraction / regulation of sodium ion transport / negative regulation of blood pressure / Ion homeostasis / response to hormone / photoreceptor inner segment / nitric oxide biosynthetic process / T-tubule / sarcoplasmic reticulum membrane / cell redox homeostasis / calyx of Held / sarcoplasmic reticulum / cell periphery / calcium channel regulator activity / establishment of localization in cell / sarcolemma / caveola / cellular response to growth factor stimulus / potassium ion transport / vasodilation / Z disc / calcium-dependent protein binding / calcium ion transport / FMN binding / flavin adenine dinucleotide binding / NADP binding / positive regulation of neuron apoptotic process / response to heat / scaffold protein binding / response to lipopolysaccharide / dendritic spine / transmembrane transporter binding / calmodulin binding / cytoskeleton / response to hypoxia / postsynaptic density / membrane raft / heme binding / synapse / positive regulation of DNA-templated transcription / perinuclear region of cytoplasm / positive regulation of transcription by RNA polymerase II / protein-containing complex / mitochondrion / nucleoplasm / nucleus / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  フーリエ合成 / 解像度: 1.93 Å フーリエ合成 / 解像度: 1.93 Å | ||||||
 データ登録者 データ登録者 | Li, H. / Poulos, T.L. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: J. Med. Chem. / 年: 2019 ジャーナル: J. Med. Chem. / 年: 2019タイトル: Optimization of Blood-Brain Barrier Permeability with Potent and Selective Human Neuronal Nitric Oxide Synthase Inhibitors Having a 2-Aminopyridine Scaffold. 著者: Do, H.T. / Li, H. / Chreifi, G. / Poulos, T.L. / Silverman, R.B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6ng2.cif.gz 6ng2.cif.gz | 369.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6ng2.ent.gz pdb6ng2.ent.gz | 300.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6ng2.json.gz 6ng2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6ng2_validation.pdf.gz 6ng2_validation.pdf.gz | 1.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6ng2_full_validation.pdf.gz 6ng2_full_validation.pdf.gz | 1.7 MB | 表示 | |
| XML形式データ |  6ng2_validation.xml.gz 6ng2_validation.xml.gz | 38 KB | 表示 | |
| CIF形式データ |  6ng2_validation.cif.gz 6ng2_validation.cif.gz | 55.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ng/6ng2 https://data.pdbj.org/pub/pdb/validation_reports/ng/6ng2 ftp://data.pdbj.org/pub/pdb/validation_reports/ng/6ng2 ftp://data.pdbj.org/pub/pdb/validation_reports/ng/6ng2 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ng1C  6ng4C  6ng5C  6ng6C  6ng7C  6ng8C  6ngaC  6ngbC  6ngcC  6ngdC  6ngeC  6ngfC  6nghC  6ngiC  6ngjC  6ngkC  6nglC  6ngmC  6ngnC  6ngpC  6ngqC  6ngrC  6ngsC  6ngtC  6nguC  6ngvC  6ngwC  6ngxC  6ngyC  6ngzC  6nh0C  6nh1C  6nh2C  6nh3C  6nh4C  6nh5C  6nh6C  6nh7C  6nh8C  6nhbC  6nhcC  6nhdC  6nheC  6nhfC  5ad7S S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 48784.496 Da / 分子数: 2 / 断片: UNP residues 302-722 / 変異: R354A,G357D / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: NOS1 / 器官: brain / プラスミド: pCWori / 発現宿主: Homo sapiens (ヒト) / 遺伝子: NOS1 / 器官: brain / プラスミド: pCWori / 発現宿主:  |
|---|
-非ポリマー , 5種, 596分子 

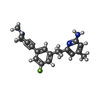






| #2: 化合物 | | #3: 化合物 | #4: 化合物 | #5: 化合物 | ChemComp-ZN / | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.74 Å3/Da / 溶媒含有率: 55.2 % / 解説: plates |
|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.2 詳細: 8% PEG3350, 35 mM citric acid, 65 mM Bis-Tris propane, 10% glycerol, 5 mM TCEP |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL14-1 / 波長: 1.195 Å / ビームライン: BL14-1 / 波長: 1.195 Å |
| 検出器 | タイプ: MARMOSAIC 325 mm CCD / 検出器: CCD / 日付: 2017年7月28日 / 詳細: mirrors |
| 放射 | モノクロメーター: double crystal Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.195 Å / 相対比: 1 |
| 反射 | 解像度: 1.93→60 Å / Num. obs: 79425 / % possible obs: 99.5 % / Observed criterion σ(I): -3 / 冗長度: 5.5 % / CC1/2: 0.998 / Rmerge(I) obs: 0.099 / Rpim(I) all: 0.066 / Rsym value: 0.099 / Net I/σ(I): 8.2 |
| 反射 シェル | 解像度: 1.93→1.99 Å / 冗長度: 5.4 % / Rmerge(I) obs: 1.703 / Mean I/σ(I) obs: 0.8 / Num. unique obs: 4494 / CC1/2: 0.436 / Rpim(I) all: 1.163 / Rsym value: 1.703 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  フーリエ合成 フーリエ合成開始モデル: PDB entry 5AD7 解像度: 1.93→37.822 Å / SU ML: 0.26 / 交差検証法: FREE R-VALUE / σ(F): 0.01 / 位相誤差: 27.75
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.93→37.822 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj









