[English] 日本語
 Yorodumi
Yorodumi- PDB-6ngq: Structure of rat neuronal nitric oxide synthase heme domain in co... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6ngq | ||||||
|---|---|---|---|---|---|---|---|
| Title | Structure of rat neuronal nitric oxide synthase heme domain in complex with 6-(3-fluoro-5-(2-((2R,4S)-4-fluoropyrrolidin-2-yl)ethyl)phenethyl)-4-methylpyridin-2-amine | ||||||
 Components Components | Nitric oxide synthase, brain | ||||||
 Keywords Keywords | oxidoreductase/oxidoreductase inhibitor / nitric oxide synthase inhibitor complex heme enzyme / OXIDOREDUCTASE / oxidoreductase-oxidoreductase inhibitor complex | ||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of hepatic stellate cell contraction / Nitric oxide stimulates guanylate cyclase / positive regulation of adenylate cyclase-activating adrenergic receptor signaling pathway / negative regulation of iron ion transmembrane transport / response to vitamin B3 / postsynaptic specialization, intracellular component / ROS and RNS production in phagocytes / azurophil granule / Ion homeostasis / synaptic signaling by nitric oxide ...negative regulation of hepatic stellate cell contraction / Nitric oxide stimulates guanylate cyclase / positive regulation of adenylate cyclase-activating adrenergic receptor signaling pathway / negative regulation of iron ion transmembrane transport / response to vitamin B3 / postsynaptic specialization, intracellular component / ROS and RNS production in phagocytes / azurophil granule / Ion homeostasis / synaptic signaling by nitric oxide / negative regulation of vasoconstriction / response to nitric oxide / positive regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / response to vitamin E / negative regulation of cytosolic calcium ion concentration / positive regulation of sodium ion transmembrane transport / peptidyl-cysteine S-nitrosylase activity / positive regulation of the force of heart contraction / cadmium ion binding / negative regulation of calcium ion transport / negative regulation of potassium ion transport / regulation of postsynaptic membrane potential / nitric oxide mediated signal transduction / sodium channel regulator activity / nitric-oxide synthase (NADPH) / regulation of neurogenesis / negative regulation of serotonin uptake / nitric-oxide synthase activity / xenobiotic catabolic process / multicellular organismal response to stress / L-arginine catabolic process / NADPH binding / postsynaptic density, intracellular component / striated muscle contraction / nitric oxide-cGMP-mediated signaling / negative regulation of blood pressure / regulation of sodium ion transport / response to hormone / nitric oxide metabolic process / behavioral response to cocaine / nitric oxide biosynthetic process / photoreceptor inner segment / cellular response to epinephrine stimulus / T-tubule / sarcoplasmic reticulum membrane / secretory granule / calyx of Held / sarcoplasmic reticulum / positive regulation of long-term synaptic potentiation / response to activity / cell periphery / response to nicotine / response to nutrient levels / phosphoprotein binding / establishment of protein localization / establishment of localization in cell / female pregnancy / cellular response to mechanical stimulus / negative regulation of insulin secretion / sarcolemma / response to peptide hormone / caveola / potassium ion transport / response to lead ion / cellular response to growth factor stimulus / response to estrogen / vasodilation / Z disc / calcium-dependent protein binding / calcium ion transport / FMN binding / NADP binding / flavin adenine dinucleotide binding / positive regulation of neuron apoptotic process / response to heat / ATPase binding / scaffold protein binding / response to ethanol / nuclear membrane / response to lipopolysaccharide / dendritic spine / negative regulation of neuron apoptotic process / RNA polymerase II-specific DNA-binding transcription factor binding / transmembrane transporter binding / response to hypoxia / mitochondrial outer membrane / cytoskeleton / calmodulin binding / postsynaptic density / membrane raft / negative regulation of cell population proliferation / heme binding / dendrite / synapse / negative regulation of apoptotic process / positive regulation of DNA-templated transcription / perinuclear region of cytoplasm / glutamatergic synapse / enzyme binding / positive regulation of transcription by RNA polymerase II Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  FOURIER SYNTHESIS / Resolution: 1.95 Å FOURIER SYNTHESIS / Resolution: 1.95 Å | ||||||
 Authors Authors | Li, H. / Poulos, T.L. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: J. Med. Chem. / Year: 2019 Journal: J. Med. Chem. / Year: 2019Title: Optimization of Blood-Brain Barrier Permeability with Potent and Selective Human Neuronal Nitric Oxide Synthase Inhibitors Having a 2-Aminopyridine Scaffold. Authors: Do, H.T. / Li, H. / Chreifi, G. / Poulos, T.L. / Silverman, R.B. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6ngq.cif.gz 6ngq.cif.gz | 365.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6ngq.ent.gz pdb6ngq.ent.gz | 295.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6ngq.json.gz 6ngq.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ng/6ngq https://data.pdbj.org/pub/pdb/validation_reports/ng/6ngq ftp://data.pdbj.org/pub/pdb/validation_reports/ng/6ngq ftp://data.pdbj.org/pub/pdb/validation_reports/ng/6ngq | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6ng1C  6ng2C  6ng4C  6ng5C  6ng6C  6ng7C  6ng8C  6ngaC  6ngbC  6ngcC  6ngdC  6ngeC  6ngfC  6nghC  6ngiC  6ngjC  6ngkC  6nglC  6ngmC  6ngnC  6ngpC  6ngrC  6ngsC  6ngtC  6nguC  6ngvC  6ngwC  6ngxC  6ngyC  6ngzC  6nh0C  6nh1C  6nh2C  6nh3C  6nh4C  6nh5C  6nh6C  6nh7C  6nh8C  6nhbC  6nhcC  6nhdC  6nheC  6nhfC  1om4S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 48812.527 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|
-Non-polymers , 6 types, 467 molecules 

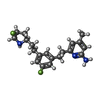








| #2: Chemical | | #3: Chemical | #4: Chemical | #5: Chemical | #6: Chemical | ChemComp-ZN / | #7: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.48 Å3/Da / Density % sol: 50.4 % / Description: bricks |
|---|---|
| Crystal grow | Temperature: 277 K / Method: vapor diffusion, sitting drop / pH: 5.8 Details: 20-24% PEG3350, 0.1M MES 0.14-0.20M AMMONIUM ACETATE, 10% ETHYLENE GLYCOL, 30uM SDS, 5 mM GSH |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 8.2.1 / Wavelength: 1 Å / Beamline: 8.2.1 / Wavelength: 1 Å |
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Apr 15, 2017 |
| Radiation | Monochromator: graphite / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 1.95→82 Å / Num. obs: 69734 / % possible obs: 99.8 % / Observed criterion σ(I): -3 / Redundancy: 4.6 % / CC1/2: 0.994 / Rmerge(I) obs: 0.124 / Rpim(I) all: 0.094 / Rsym value: 0.124 / Net I/σ(I): 5.7 |
| Reflection shell | Resolution: 1.95→2.02 Å / Redundancy: 4.4 % / Rmerge(I) obs: 2.226 / Num. unique obs: 4473 / CC1/2: 0.501 / Rpim(I) all: 1.765 / Rsym value: 2.226 / % possible all: 100 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  FOURIER SYNTHESIS FOURIER SYNTHESISStarting model: 1OM4 Resolution: 1.95→81.79 Å / SU ML: 0.31 / Cross valid method: FREE R-VALUE / σ(F): 0.03 / Phase error: 31.29
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.95→81.79 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller






















 PDBj
PDBj








