+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6myp | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Avian mitochondrial complex II with TTFA (thenoyltrifluoroacetone) bound | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | OXIDOREDUCTASE/OXIDOREDUCTASE INHIBITOR / Complex II / membrane protein / heme protein / OXIDOREDUCTASE-OXIDOREDUCTASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / Citric acid cycle (TCA cycle) / The tricarboxylic acid cycle / succinate dehydrogenase activity / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; キノンあるいはその類似化合物を用いる / succinate metabolic process / respiratory chain complex II (succinate dehydrogenase) / mitochondrial electron transport, succinate to ubiquinone / succinate dehydrogenase (quinone) activity / succinate dehydrogenase ...: / Citric acid cycle (TCA cycle) / The tricarboxylic acid cycle / succinate dehydrogenase activity / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; キノンあるいはその類似化合物を用いる / succinate metabolic process / respiratory chain complex II (succinate dehydrogenase) / mitochondrial electron transport, succinate to ubiquinone / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / 3 iron, 4 sulfur cluster binding / ubiquinone binding / tricarboxylic acid cycle / aerobic respiration / respiratory electron transport chain / mitochondrial membrane / 2 iron, 2 sulfur cluster binding / flavin adenine dinucleotide binding / 4 iron, 4 sulfur cluster binding / electron transfer activity / mitochondrial inner membrane / heme binding / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å | ||||||
 データ登録者 データ登録者 | Berry, E.A. / Huang, L.-S. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Biochim Biophys Acta Proteins Proteom / 年: 2021 ジャーナル: Biochim Biophys Acta Proteins Proteom / 年: 2021タイトル: Crystallographic investigation of the ubiquinone binding site of respiratory Complex II and its inhibitors. 著者: Huang, L.S. / Lummen, P. / Berry, E.A. #1:  ジャーナル: J. Biol. Chem. / 年: 2006 ジャーナル: J. Biol. Chem. / 年: 2006タイトル: 3-nitropropionic acid is a suicide inhibitor of mitochondrial respiration that, upon oxidation by complex II, forms a covalent adduct with a catalytic base arginine in the active site of the enzyme. 著者: Huang, L.S. / Sun, G. / Cobessi, D. / Wang, A.C. / Shen, J.T. / Tung, E.Y. / Anderson, V.E. / Berry, E.A. #2:  ジャーナル: Biochim. Biophys. Acta / 年: 2006 ジャーナル: Biochim. Biophys. Acta / 年: 2006タイトル: Crystallographic studies of the binding of ligands to the dicarboxylate site of Complex II, and the identity of the ligand in the "oxaloacetate-inhibited" state. 著者: Huang, L.S. / Shen, J.T. / Wang, A.C. / Berry, E.A. #3: ジャーナル: Acta Crystallogr. D Biol. Crystallogr. / 年: 2005 タイトル: Crystallization of mitochondrial respiratory complex II from chicken heart: a membrane-protein complex diffracting to 2.0 A. 著者: Huang, L.S. / Borders, T.M. / Shen, J.T. / Wang, C.J. / Berry, E.A. #4:  ジャーナル: Cell / 年: 2005 ジャーナル: Cell / 年: 2005タイトル: Crystal structure of mitochondrial respiratory membrane protein complex II. 著者: Sun, F. / Huo, X. / Zhai, Y. / Wang, A. / Xu, J. / Su, D. / Bartlam, M. / Rao, Z. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6myp.cif.gz 6myp.cif.gz | 493.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6myp.ent.gz pdb6myp.ent.gz | 397.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6myp.json.gz 6myp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6myp_validation.pdf.gz 6myp_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6myp_full_validation.pdf.gz 6myp_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  6myp_validation.xml.gz 6myp_validation.xml.gz | 46.4 KB | 表示 | |
| CIF形式データ |  6myp_validation.cif.gz 6myp_validation.cif.gz | 64.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/my/6myp https://data.pdbj.org/pub/pdb/validation_reports/my/6myp ftp://data.pdbj.org/pub/pdb/validation_reports/my/6myp ftp://data.pdbj.org/pub/pdb/validation_reports/my/6myp | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Succinate dehydrogenase [ubiquinone] ... , 3種, 3分子 ABD
| #1: タンパク質 | 分子量: 68256.922 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  参照: UniProt: F1NPJ4, UniProt: Q9YHT1*PLUS, succinate dehydrogenase |
|---|---|
| #2: タンパク質 | 分子量: 28685.221 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #4: タンパク質 | 分子量: 10971.604 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
-タンパク質 , 1種, 1分子 C
| #3: タンパク質 | 分子量: 15391.153 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  参照: UniProt: D0VWW3, UniProt: A0A3Q2U2Y6*PLUS, succinate dehydrogenase |
|---|
-非ポリマー , 15種, 513分子 








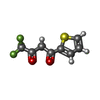
















| #5: 化合物 | ChemComp-FAD / | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #6: 化合物 | ChemComp-OAA / | ||||||||||||||||
| #7: 化合物 | ChemComp-K / | ||||||||||||||||
| #8: 化合物 | ChemComp-MG / | ||||||||||||||||
| #9: 化合物 | ChemComp-BCT / | ||||||||||||||||
| #10: 化合物 | ChemComp-PEG / | ||||||||||||||||
| #11: 化合物 | ChemComp-UNL / 分子量: 106.120 Da / 分子数: 63 / 由来タイプ: 合成 #12: 化合物 | ChemComp-FES / | #13: 化合物 | ChemComp-SF4 / | #14: 化合物 | ChemComp-F3S / | #15: 化合物 | ChemComp-TTF / | #16: 化合物 | ChemComp-HEM / | #17: 化合物 | ChemComp-UMQ / | #18: 化合物 | ChemComp-3PE / | #19: 水 | ChemComp-HOH / | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.47 Å3/Da / 溶媒含有率: 64.55 % |
|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 48 g/L PEG3350, 15 mL/L PEG400, 24 mL/L isopropanol, 0.05 M HEPES sodium, 0.01 M Tris-HCl, 1.6 mM manganese chloride, 0.64 mM magnesium chloride, 1.5 mM sodium azide, 0.25 mM sodium EDTA, 10 ...詳細: 48 g/L PEG3350, 15 mL/L PEG400, 24 mL/L isopropanol, 0.05 M HEPES sodium, 0.01 M Tris-HCl, 1.6 mM manganese chloride, 0.64 mM magnesium chloride, 1.5 mM sodium azide, 0.25 mM sodium EDTA, 10 g/L octyl beta-D-glucoside, undecyl-beta-D-maltoside, TTFA |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 8.2.2 / 波長: 0.9743 Å / ビームライン: 8.2.2 / 波長: 0.9743 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2004年10月9日 |
| 放射 | モノクロメーター: double crystal Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9743 Å / 相対比: 1 |
| 反射 | 解像度: 2.1→50.36 Å / Num. obs: 100834 / % possible obs: 99.6 % / Observed criterion σ(I): -3 / 冗長度: 4.7 % / Biso Wilson estimate: 34.29 Å2 / Rmerge(I) obs: 0.114 / Net I/σ(I): 6.9 |
| 反射 シェル | 解像度: 2.1→2.16 Å / 冗長度: 4.56 % / Rmerge(I) obs: 0.636 / Mean I/σ(I) obs: 2.2 / % possible all: 99.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1YQ3 解像度: 2.1→50.36 Å / SU ML: 0.32 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 30.97
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 51.7618 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→50.36 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: 20.3751 Å / Origin y: 18.9961 Å / Origin z: 110.21 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: ALL |
 ムービー
ムービー コントローラー
コントローラー






























 PDBj
PDBj




























