[English] 日本語
 Yorodumi
Yorodumi- PDB-6lwd: Crystal structure of human NEIL1(P2G, E3Q, R242) bound to duplex ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6lwd | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of human NEIL1(P2G, E3Q, R242) bound to duplex DNA containing spiroiminodihydantoin (Sp) | |||||||||
 Components Components |
| |||||||||
 Keywords Keywords | DNA BINDING PROTEIN/DNA / Base lesion / Oxidative DNA damage / DNA repair / DNA BINDING PROTEIN-DNA complex | |||||||||
| Function / homology |  Function and homology information Function and homology information: / Defective Base Excision Repair Associated with NEIL1 / depyrimidination / DNA N-glycosylase activity / hydrolase activity, acting on glycosyl bonds / Hydrolases; Glycosylases; Hydrolysing N-glycosyl compounds / APEX1-Independent Resolution of AP Sites via the Single Nucleotide Replacement Pathway / base-excision repair, gap-filling / DNA-(apurinic or apyrimidinic site) endonuclease activity / Recognition and association of DNA glycosylase with site containing an affected pyrimidine ...: / Defective Base Excision Repair Associated with NEIL1 / depyrimidination / DNA N-glycosylase activity / hydrolase activity, acting on glycosyl bonds / Hydrolases; Glycosylases; Hydrolysing N-glycosyl compounds / APEX1-Independent Resolution of AP Sites via the Single Nucleotide Replacement Pathway / base-excision repair, gap-filling / DNA-(apurinic or apyrimidinic site) endonuclease activity / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / base-excision repair / chromosome / response to oxidative stress / damaged DNA binding / centrosome / zinc ion binding / nucleoplasm / nucleus / cytosol / cytoplasm Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.41 Å MOLECULAR REPLACEMENT / Resolution: 2.41 Å | |||||||||
 Authors Authors | Liu, M.H. / Zhang, J. / Zhu, C.X. / Zhang, X.X. / Gao, Y.Q. / Yi, C.Q. | |||||||||
| Funding support |  China, 2items China, 2items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2021 Journal: Nat Commun / Year: 2021Title: DNA repair glycosylase hNEIL1 triages damaged bases via competing interaction modes. Authors: Liu, M. / Zhang, J. / Zhu, C. / Zhang, X. / Xiao, W. / Yan, Y. / Liu, L. / Zeng, H. / Gao, Y.Q. / Yi, C. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6lwd.cif.gz 6lwd.cif.gz | 401.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6lwd.ent.gz pdb6lwd.ent.gz | 322.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6lwd.json.gz 6lwd.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/lw/6lwd https://data.pdbj.org/pub/pdb/validation_reports/lw/6lwd ftp://data.pdbj.org/pub/pdb/validation_reports/lw/6lwd ftp://data.pdbj.org/pub/pdb/validation_reports/lw/6lwd | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6lwaC  6lwbC  6lwcC  6lwfC  6lwgC  6lwhC  6lwiC  6lwjC  6lwkC  6lwlC  6lwmC  6lwnC  6lwoC  6lwpC  6lwqC  6lwrC  5ityS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 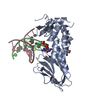
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 33288.184 Da / Num. of mol.: 3 / Mutation: P2G, E3Q, K242 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: NEIL1 / Production host: Homo sapiens (human) / Gene: NEIL1 / Production host:  References: UniProt: Q96FI4, Hydrolases; Glycosylases; Hydrolysing N-glycosyl compounds, DNA-(apurinic or apyrimidinic site) lyase #2: DNA chain | Mass: 3959.560 Da / Num. of mol.: 3 / Source method: obtained synthetically / Details: Oligo DNA containing Sp / Source: (synth.)  #3: DNA chain | Mass: 4016.623 Da / Num. of mol.: 3 / Source method: obtained synthetically / Details: Complementary strand / Source: (synth.)  #4: Chemical | ChemComp-GOL / | #5: Water | ChemComp-HOH / | Has ligand of interest | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.75 Å3/Da / Density % sol: 55.29 % |
|---|---|
| Crystal grow | Temperature: 277.15 K / Method: vapor diffusion, hanging drop / pH: 6.5 Details: 0.1 M cacodylic acid (pH 6.5), 0.1 M NaCl, 0.05 M MgCl2 and 18% (w/v) PEG 8000 |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SSRF SSRF  / Beamline: BL17U1 / Wavelength: 0.9793 Å / Beamline: BL17U1 / Wavelength: 0.9793 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: MAR CCD 130 mm / Detector: CCD / Date: Jan 14, 2017 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9793 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 2.4→50 Å / Num. obs: 53244 / % possible obs: 99.6 % / Redundancy: 5.3 % / Rmerge(I) obs: 0.082 / Rpim(I) all: 0.04 / Rrim(I) all: 0.091 / Χ2: 1.522 / Net I/σ(I): 11.4 / Num. measured all: 284513 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5ITY Resolution: 2.41→42.49 Å / Cor.coef. Fo:Fc: 0.945 / Cor.coef. Fo:Fc free: 0.924 / SU B: 16.123 / SU ML: 0.186 / Cross valid method: THROUGHOUT / σ(F): 0 / ESU R: 0.322 / ESU R Free: 0.243 / Stereochemistry target values: MAXIMUM LIKELIHOOD Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : WITH TLS ADDED
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 167.02 Å2 / Biso mean: 72.969 Å2 / Biso min: 33.33 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.41→42.49 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.41→2.468 Å / Rfactor Rfree error: 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller



















 PDBj
PDBj









































