+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6lr2 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | SOLUTION STRUCTURE OF THE YTH DOMAIN IN YTH DOMAIN-2 CONTAINING PROTEIN 2 | ||||||
 要素 要素 | YTH domain containing protein 2 (YTHDC2) | ||||||
 キーワード キーワード | RNA BINDING PROTEIN / MEIOSIS RELATED PROTEIN / Structural Genomics / PSI-2 / Protein Structure Initiative / RIKEN Structural Genomics/Proteomics Initiative / RSGI | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報germline cell cycle switching, mitotic to meiotic cell cycle / ribonucleoprotein granule / host-mediated activation of viral genome replication / 3'-5' RNA helicase activity / N6-methyladenosine-containing RNA reader activity / oocyte development / ATP-dependent activity, acting on RNA / RNA polymerase binding / response to tumor necrosis factor / spermatid development ...germline cell cycle switching, mitotic to meiotic cell cycle / ribonucleoprotein granule / host-mediated activation of viral genome replication / 3'-5' RNA helicase activity / N6-methyladenosine-containing RNA reader activity / oocyte development / ATP-dependent activity, acting on RNA / RNA polymerase binding / response to tumor necrosis factor / spermatid development / response to interleukin-1 / meiotic cell cycle / helicase activity / RNA helicase / perinuclear region of cytoplasm / endoplasmic reticulum / ATP hydrolysis activity / RNA binding / ATP binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Muto, Y. / Kobayashi, N. / Yokoyama, S. / RIKEN Structural Genomics/Proteomics Initiative (RSGI) | ||||||
 引用 引用 |  ジャーナル: TO BE PUBLISHED ジャーナル: TO BE PUBLISHEDタイトル: SOLUTION STRUCTURE OF THE YTH DOMAIN IN YTH DOMAIN-CONTAINING PROTEIN 2 著者: Endo, R. / He, F. / Inoue, M. / Muto, Y. / Kigawa, T. / Shirouzu, M. / Yokoyama, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6lr2.cif.gz 6lr2.cif.gz | 869.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6lr2.ent.gz pdb6lr2.ent.gz | 729.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6lr2.json.gz 6lr2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6lr2_validation.pdf.gz 6lr2_validation.pdf.gz | 548.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6lr2_full_validation.pdf.gz 6lr2_full_validation.pdf.gz | 683.6 KB | 表示 | |
| XML形式データ |  6lr2_validation.xml.gz 6lr2_validation.xml.gz | 49.6 KB | 表示 | |
| CIF形式データ |  6lr2_validation.cif.gz 6lr2_validation.cif.gz | 81.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lr/6lr2 https://data.pdbj.org/pub/pdb/validation_reports/lr/6lr2 ftp://data.pdbj.org/pub/pdb/validation_reports/lr/6lr2 ftp://data.pdbj.org/pub/pdb/validation_reports/lr/6lr2 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 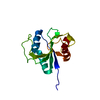
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 16138.065 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / プラスミド: P060508-16 Homo sapiens (ヒト) / プラスミド: P060508-16発現宿主: Cell-free gateway cloning vector N-term 8xHis eGFP pCellFree_G03 (その他) 参照: UniProt: Q6ZMY0, UniProt: Q9H6S0*PLUS |
|---|---|
| 配列の詳細 | Authors state that 6L2R has a mutation, L129Q based rs1132529 in dbSNP database. |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験 | Sample state: isotropic / タイプ: 3D 13C,15N-SEPARATED NOESY SPECTRA |
- 試料調製
試料調製
| 詳細 | タイプ: solution 内容: 1.0 mg/mL [U-99% 13C; U-99% 15N] helicase, 90% H2O/10% D2O 詳細: 1mM D-DTT;0.02% NaN3 were used to keep the protein condition Label: 13C, 15N_sample / 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料 | 濃度: 1.0 mg/mL / 構成要素: helicase / Isotopic labeling: [U-99% 13C; U-99% 15N] |
| 試料状態 | 詳細: 20mM D-Tris-HCL (PH7.0); 100mM NaCl; 1mM D-DTT;0.02% NaN3 イオン強度: 100 mM / Label: condition_1 / pH: 7 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AVANCE / 製造業者: Bruker / モデル: AVANCE / 磁場強度: 800 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: the structures are based on 3191 NOE-derived distance contrrains, 71 main chain dihedral angle constraints based on TALOS program and 51 side chain dihedral constraints based on NOE pattern. | ||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with favorable non-bond energy 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
 gel filtration
gel filtration