+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6lkn | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of ATP11C-CDC50A in PtdSer-bound E2P state | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | MEMBRANE PROTEIN / P-type ATPase / flippase / apoptosis / phosphatidylserine | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of phospholipid translocation / aminophospholipid transport / aminophospholipid flippase activity / phosphatidylserine flippase activity / protein localization to endosome / ATPase-coupled intramembrane lipid transporter activity / phospholipid-translocating ATPase complex / phosphatidylserine floppase activity / positive regulation of protein exit from endoplasmic reticulum / xenobiotic transmembrane transport ...positive regulation of phospholipid translocation / aminophospholipid transport / aminophospholipid flippase activity / phosphatidylserine flippase activity / protein localization to endosome / ATPase-coupled intramembrane lipid transporter activity / phospholipid-translocating ATPase complex / phosphatidylserine floppase activity / positive regulation of protein exit from endoplasmic reticulum / xenobiotic transmembrane transport / phosphatidylethanolamine flippase activity / P-type phospholipid transporter / phospholipid translocation / azurophil granule membrane / transport vesicle membrane / Ion transport by P-type ATPases / specific granule membrane / recycling endosome / positive regulation of neuron projection development / recycling endosome membrane / late endosome membrane / early endosome membrane / monoatomic ion transmembrane transport / apical plasma membrane / lysosomal membrane / Neutrophil degranulation / endoplasmic reticulum membrane / structural molecule activity / Golgi apparatus / magnesium ion binding / endoplasmic reticulum / ATP hydrolysis activity / ATP binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.9 Å 分子置換 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Abe, K. / Irie, K. / Nakanishi, H. / Hasegawa, K. | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2020 ジャーナル: J.Biol.Chem. / 年: 2020タイトル: Crystal structure of a human plasma membrane phospholipid flippase. 著者: Nakanishi, H. / Irie, K. / Segawa, K. / Hasegawa, K. / Fujiyoshi, Y. / Nagata, S. / Abe, K. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6lkn.cif.gz 6lkn.cif.gz | 2.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6lkn.ent.gz pdb6lkn.ent.gz | 1.9 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6lkn.json.gz 6lkn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6lkn_validation.pdf.gz 6lkn_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6lkn_full_validation.pdf.gz 6lkn_full_validation.pdf.gz | 2 MB | 表示 | |
| XML形式データ |  6lkn_validation.xml.gz 6lkn_validation.xml.gz | 215.1 KB | 表示 | |
| CIF形式データ |  6lkn_validation.cif.gz 6lkn_validation.cif.gz | 280.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lk/6lkn https://data.pdbj.org/pub/pdb/validation_reports/lk/6lkn ftp://data.pdbj.org/pub/pdb/validation_reports/lk/6lkn ftp://data.pdbj.org/pub/pdb/validation_reports/lk/6lkn | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6k7lS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| 4 | 
| ||||||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 8分子 AEIMCFJN
| #1: タンパク質 | 分子量: 129710.633 Da / 分子数: 4 / 変異: C111W / 由来タイプ: 組換発現 詳細: Amino acid 410 in PDB is BFD, which is BeF-bound Asp. 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: ATP11C, ATPIG, ATPIQ / 細胞株 (発現宿主): Expi293 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: ATP11C, ATPIG, ATPIQ / 細胞株 (発現宿主): Expi293 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q8NB49, P-type phospholipid transporter Homo sapiens (ヒト) / 参照: UniProt: Q8NB49, P-type phospholipid transporter#2: タンパク質 | 分子量: 40840.684 Da / 分子数: 4 / 変異: N190Q, S292W / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: TMEM30A, C6orf67, CDC50A / 細胞株 (発現宿主): Expi293 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TMEM30A, C6orf67, CDC50A / 細胞株 (発現宿主): Expi293 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q9NV96 Homo sapiens (ヒト) / 参照: UniProt: Q9NV96 |
|---|
-糖 , 2種, 20分子 
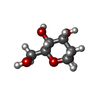

| #5: 糖 | ChemComp-NAG / #6: 糖 | ChemComp-AH2 / |
|---|
-非ポリマー , 3種, 19分子 




| #3: 化合物 | ChemComp-P5S / #4: 化合物 | ChemComp-MG / #7: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.23 Å3/Da / 溶媒含有率: 70.9 % |
|---|---|
| 結晶化 | 温度: 293.15 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.5 詳細: 14-15% PEG4000, 10% glycerol, 5 mM beta-mercaptoethanol, 0.4 M MgSO4 |
-データ収集
| 回折 | 平均測定温度: 80 K / Ambient temp details: LN2 / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL41XU / 波長: 1 Å / ビームライン: BL41XU / 波長: 1 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2019年4月19日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 3.9→50 Å / Num. obs: 106458 / % possible obs: 71.32 % / 冗長度: 776.9 % / Biso Wilson estimate: 98.24 Å2 / CC1/2: 0.92 / Rmerge(I) obs: 1.141 / Rpim(I) all: 0.0419 / Net I/σ(I): 15.88 |
| 反射 シェル | 解像度: 3.9→4 Å / 冗長度: 698.5 % / Num. unique obs: 1223 / CC1/2: 0.864 / % possible all: 11.64 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 6k7l 解像度: 3.9→49.97 Å / SU ML: 0.6547 / 交差検証法: FREE R-VALUE / σ(F): 2.07 / 位相誤差: 38.7776 詳細: Diffractions were strongly anisorlopic. We merged 1588 crystal data to improve data quality.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 161.97 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.9→49.97 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -25.8822080037 Å / Origin y: 100.493022409 Å / Origin z: -125.137573873 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj









