[English] 日本語
 Yorodumi
Yorodumi- PDB-6l6y: Crystal Structure of Pluripotency Reprogramming Factor Sox17 muta... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6l6y | ||||||
|---|---|---|---|---|---|---|---|
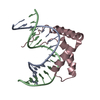
| Title | Crystal Structure of Pluripotency Reprogramming Factor Sox17 mutant (Sox17EK) HMG Domain bound to DNA | ||||||
 Components Components |
| ||||||
 Keywords Keywords | DNA BINDING PROTEIN/DNA / TRANSCRIPTION / HMG / Stem Cell Transcription Factor / Pluripotency Reprogramming Factor / DNA BINDING PROTEIN-DNA / TRANSCRIPTION complex | ||||||
| Function / homology |  Function and homology information Function and homology informationcardiogenic plate morphogenesis / endodermal cell fate determination / regulation of cardiac cell fate specification / stem cell fate specification / endocardium formation / common bile duct development / endodermal digestive tract morphogenesis / gallbladder development / ureter development / inner cell mass cellular morphogenesis ...cardiogenic plate morphogenesis / endodermal cell fate determination / regulation of cardiac cell fate specification / stem cell fate specification / endocardium formation / common bile duct development / endodermal digestive tract morphogenesis / gallbladder development / ureter development / inner cell mass cellular morphogenesis / cardiac cell fate determination / endodermal cell fate specification / regulation of stem cell division / Deactivation of the beta-catenin transactivating complex / cell migration involved in gastrulation / endocardial cell differentiation / rostrocaudal neural tube patterning / positive regulation of endodermal cell differentiation / embryonic foregut morphogenesis / positive regulation of stem cell differentiation / regulation of stem cell proliferation / endoderm development / response to alkaloid / metanephros development / embryonic heart tube morphogenesis / embryonic heart tube development / signal transduction involved in regulation of gene expression / heart looping / endodermal cell differentiation / regulation of cell differentiation / negative regulation of Wnt signaling pathway / outflow tract morphogenesis / regulation of embryonic development / embryonic organ development / vasculogenesis / gastrulation / cellular response to leukemia inhibitory factor / stem cell differentiation / negative regulation of canonical Wnt signaling pathway / negative regulation of cell growth / beta-catenin binding / protein destabilization / Wnt signaling pathway / positive regulation of protein catabolic process / heart development / DNA-binding transcription activator activity, RNA polymerase II-specific / angiogenesis / spermatogenesis / transcription regulator complex / gene expression / sequence-specific DNA binding / RNA polymerase II-specific DNA-binding transcription factor binding / DNA-binding transcription factor activity, RNA polymerase II-specific / transcription cis-regulatory region binding / protein stabilization / DNA-binding transcription factor activity / positive regulation of gene expression / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / DNA binding / nucleoplasm / nucleus Similarity search - Function | ||||||
| Biological species |   Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / Resolution: 3 Å MOLECULAR REPLACEMENT / Resolution: 3 Å | ||||||
 Authors Authors | Balasubramanian, M. / Kolatkar, P.R. | ||||||
| Funding support | Qatar, 1items
| ||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Crystal Structure of Pluripotency Reprogramming Factor Sox17 mutant (Sox17EK) HMG Domain bound to DNA Authors: Balasubramanian, M. / Kolatkar, P.R. #1:  Journal: Bioinformatics / Year: 2019 Journal: Bioinformatics / Year: 2019Title: DeepCrystal: a deep learning framework for sequence-based protein crystallization prediction. Authors: Elbasir, A. / Moovarkumudalvan, B. / Kunji, K. / Kolatkar, P.R. / Mall, R. / Bensmail, H. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6l6y.cif.gz 6l6y.cif.gz | 179.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6l6y.ent.gz pdb6l6y.ent.gz | 115.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6l6y.json.gz 6l6y.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/l6/6l6y https://data.pdbj.org/pub/pdb/validation_reports/l6/6l6y ftp://data.pdbj.org/pub/pdb/validation_reports/l6/6l6y ftp://data.pdbj.org/pub/pdb/validation_reports/l6/6l6y | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  3f27S S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| Unit cell |
|
- Components
Components
| #1: DNA chain | Mass: 4871.149 Da / Num. of mol.: 2 / Source method: obtained synthetically / Source: (synth.)  Homo sapiens (human) Homo sapiens (human)#2: DNA chain | Mass: 4925.233 Da / Num. of mol.: 2 / Source method: obtained synthetically / Source: (synth.)  Homo sapiens (human) Homo sapiens (human)#3: Protein | Mass: 10084.753 Da / Num. of mol.: 2 / Mutation: E122K Source method: isolated from a genetically manipulated source Source: (gene. exp.)   #4: Chemical | ChemComp-SO4 / | #5: Water | ChemComp-HOH / | Has ligand of interest | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.68 Å3/Da / Density % sol: 54.1 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, hanging drop / pH: 8.6 Details: 0.2 M Ammonium Sulfate, 30% PEG 4000, 0.1 M Tris pH 8.6 |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source: SEALED TUBE / Type: BRUKER D8 QUEST / Details: Bruker D8 Venture / Wavelength: 1.5418 Å |
| Detector | Type: BRUKER PHOTON 100 / Detector: CMOS / Date: Jul 12, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.5418 Å / Relative weight: 1 |
| Reflection | Resolution: 2.99→20.97 Å / Num. obs: 16478 / % possible obs: 99.1 % / Redundancy: 9.61 % / Biso Wilson estimate: 32.63 Å2 / Rmerge(I) obs: 0.17 / Net I/σ(I): 11.02 |
| Reflection shell | Resolution: 2.99→3.09 Å / Redundancy: 7.04 % / Rmerge(I) obs: 0.45 / Mean I/σ(I) obs: 2.74 / Num. unique obs: 839 / % possible all: 95.3 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3F27 Resolution: 3→20.97 Å / SU ML: 0.3848 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 26.8894 Details: SF FILE CONTAINS FRIEDEL PAIRS UNDER I_MINUS AND I_PLUS COLUMNS.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 47.15 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 3→20.97 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller









 PDBj
PDBj










































