| 登録情報 | データベース: PDB / ID: 6kz1
|
|---|
| タイトル | Complex structure of Whirlin and Myosin XVa |
|---|
 要素 要素 | |
|---|
 キーワード キーワード | MOTOR PROTEIN / Whirlin / Myosin XVa / complex structure |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
paranodal junction maintenance / microfilament motor activity => GO:0000146 / periciliary membrane compartment / USH2 complex / stereocilia ankle link / actin-based cell projection / inner ear receptor cell differentiation / stereocilia ankle link complex / sensory perception of light stimulus / cerebellar Purkinje cell layer formation ...paranodal junction maintenance / microfilament motor activity => GO:0000146 / periciliary membrane compartment / USH2 complex / stereocilia ankle link / actin-based cell projection / inner ear receptor cell differentiation / stereocilia ankle link complex / sensory perception of light stimulus / cerebellar Purkinje cell layer formation / photoreceptor connecting cilium / stereocilium tip / inner ear receptor cell stereocilium organization / detection of mechanical stimulus involved in sensory perception of sound / stereocilium / vesicle transport along actin filament / retina homeostasis / auditory receptor cell stereocilium organization / myosin complex / Sensory processing of sound by outer hair cells of the cochlea / Sensory processing of sound by inner hair cells of the cochlea / microfilament motor activity / photoreceptor inner segment / ciliary basal body / establishment of localization in cell / actin filament / actin filament organization / sensory perception of sound / establishment of protein localization / cilium / actin filament binding / actin cytoskeleton / growth cone / vesicle / calmodulin binding / synapse / positive regulation of gene expression / extracellular exosome / ATP binding / identical protein binding / plasma membrane / cytoplasm類似検索 - 分子機能 Unconventional myosin-XV / Whirlin / : / Class XV myosin, motor domain / Myosin-XV, FERM domain C-lobe / MyTH4 domain / MyTH4 domain superfamily / MyTH4 domain / MyTH4 domain profile. / Domain in Myosin and Kinesin Tails ...Unconventional myosin-XV / Whirlin / : / Class XV myosin, motor domain / Myosin-XV, FERM domain C-lobe / MyTH4 domain / MyTH4 domain superfamily / MyTH4 domain / MyTH4 domain profile. / Domain in Myosin and Kinesin Tails / IQ calmodulin-binding motif / Variant SH3 domain / Short calmodulin-binding motif containing conserved Ile and Gln residues. / Myosin head, motor domain / Myosin head (motor domain) / Myosin motor domain profile. / Myosin. Large ATPases. / IQ motif profile. / IQ motif, EF-hand binding site / FERM central domain / FERM superfamily, second domain / FERM domain / FERM domain profile. / Band 4.1 domain / Band 4.1 homologues / Kinesin motor domain superfamily / PDZ domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Src homology 3 domains / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. / SH3 domain / PH-like domain superfamily / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.694 Å 分子置換 / 解像度: 1.694 Å |
|---|
 データ登録者 データ登録者 | Lin, L. / Wang, M. / Shi, Y. / Zhu, J. / Zhang, R. |
|---|
 引用 引用 |  ジャーナル: Cell Rep / 年: 2021 ジャーナル: Cell Rep / 年: 2021
タイトル: Phase separation-mediated condensation of Whirlin-Myo15-Eps8 stereocilia tip complex.
著者: Lin, L. / Shi, Y. / Wang, M. / Wang, C. / Lu, Q. / Zhu, J. / Zhang, R. |
|---|
| 履歴 | | 登録 | 2019年9月22日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2020年9月23日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2021年4月7日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2023年11月22日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.694 Å
分子置換 / 解像度: 1.694 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell Rep / 年: 2021
ジャーナル: Cell Rep / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6kz1.cif.gz
6kz1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6kz1.ent.gz
pdb6kz1.ent.gz PDB形式
PDB形式 6kz1.json.gz
6kz1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6kz1_validation.pdf.gz
6kz1_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6kz1_full_validation.pdf.gz
6kz1_full_validation.pdf.gz 6kz1_validation.xml.gz
6kz1_validation.xml.gz 6kz1_validation.cif.gz
6kz1_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/kz/6kz1
https://data.pdbj.org/pub/pdb/validation_reports/kz/6kz1 ftp://data.pdbj.org/pub/pdb/validation_reports/kz/6kz1
ftp://data.pdbj.org/pub/pdb/validation_reports/kz/6kz1
 リンク
リンク 集合体
集合体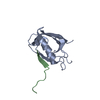

 要素
要素 Homo sapiens (ヒト) / 遺伝子: WHRN, DFNB31, KIAA1526 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: WHRN, DFNB31, KIAA1526 / 発現宿主: 
 Homo sapiens (ヒト) / 参照: UniProt: Q9UKN7*PLUS
Homo sapiens (ヒト) / 参照: UniProt: Q9UKN7*PLUS X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL41XU / 波長: 1 Å
/ ビームライン: BL41XU / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj









