Entry Database : PDB / ID : 6jbxTitle Crystal structure of Streptococcus pneumoniae FabT in complex with DNA DNA (5'-D(*AP*AP*TP*AP*GP*TP*TP*TP*GP*AP*CP*TP*GP*TP*CP*AP*AP*AP*TP*TP*AP*TP*G)-3')DNA (5'-D(*CP*AP*TP*AP*AP*TP*TP*TP*GP*AP*CP*AP*GP*TP*CP*AP*AP*AP*CP*TP*AP*TP*T)-3')Fatty acid biosynthesis transcriptional regulator Keywords / / / / Function / homology Function Domain/homology Component
/ / / / / / Biological species Streptococcus pneumoniae (bacteria)Method / / / Resolution : 2.2 Å Authors Zuo, G. / Chen, Z.P. / Li, Q. / Zhou, C.Z. Funding support Organization Grant number Country National Natural Science Foundation of China 31570728
Journal : Febs Lett. / Year : 2019Title : Structural insights into repression of the Pneumococcal fatty acid synthesis pathway by repressor FabT and co-repressor acyl-ACP.Authors : Zuo, G. / Chen, Z.P. / Jiang, Y.L. / Zhu, Z. / Ding, C. / Zhang, Z. / Chen, Y. / Zhou, C.Z. / Li, Q. History Deposition Jan 27, 2019 Deposition site / Processing site Revision 1.0 Jul 24, 2019 Provider / Type Revision 1.1 Oct 23, 2019 Group / Database references / Category / citation_authorItem _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID Revision 1.2 Mar 27, 2024 Group / Database references / Category / chem_comp_bond / database_2Item / _database_2.pdbx_database_accession
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information
 X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  SAD / Resolution: 2.2 Å
SAD / Resolution: 2.2 Å  Authors
Authors China, 1items
China, 1items  Citation
Citation Journal: Febs Lett. / Year: 2019
Journal: Febs Lett. / Year: 2019 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 6jbx.cif.gz
6jbx.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb6jbx.ent.gz
pdb6jbx.ent.gz PDB format
PDB format 6jbx.json.gz
6jbx.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/jb/6jbx
https://data.pdbj.org/pub/pdb/validation_reports/jb/6jbx ftp://data.pdbj.org/pub/pdb/validation_reports/jb/6jbx
ftp://data.pdbj.org/pub/pdb/validation_reports/jb/6jbx Links
Links Assembly
Assembly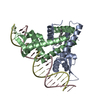
 Components
Components



 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  SSRF
SSRF  / Beamline: BL17U / Wavelength: 0.97918 Å
/ Beamline: BL17U / Wavelength: 0.97918 Å Processing
Processing SAD / Resolution: 2.2→35.96 Å / Cor.coef. Fo:Fc: 0.958 / Cor.coef. Fo:Fc free: 0.944 / SU B: 5.176 / SU ML: 0.128 / Cross valid method: THROUGHOUT / ESU R: 0.199 / ESU R Free: 0.17 / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
SAD / Resolution: 2.2→35.96 Å / Cor.coef. Fo:Fc: 0.958 / Cor.coef. Fo:Fc free: 0.944 / SU B: 5.176 / SU ML: 0.128 / Cross valid method: THROUGHOUT / ESU R: 0.199 / ESU R Free: 0.17 / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS Movie
Movie Controller
Controller












 PDBj
PDBj








































