| 登録情報 | データベース: PDB / ID: 6hq1
|
|---|
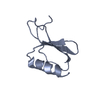
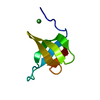
| タイトル | Solution structure of the globular domain from human histone H1.0 |
|---|
 要素 要素 | Histone H1.0 |
|---|
 キーワード キーワード | DNA BINDING PROTEIN / alpha-helical / nucleosome assembly |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of transcription regulatory region DNA binding / negative regulation of DNA recombination / Apoptosis induced DNA fragmentation / chromosome condensation / nucleosomal DNA binding / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / minor groove of adenine-thymine-rich DNA binding / nucleosome binding / transcription repressor complex / euchromatin ...positive regulation of transcription regulatory region DNA binding / negative regulation of DNA recombination / Apoptosis induced DNA fragmentation / chromosome condensation / nucleosomal DNA binding / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / minor groove of adenine-thymine-rich DNA binding / nucleosome binding / transcription repressor complex / euchromatin / chromatin DNA binding / structural constituent of chromatin / heterochromatin formation / nucleosome / nucleosome assembly / actin cytoskeleton / double-stranded DNA binding / nuclear body / chromatin / Golgi apparatus / RNA binding / nucleoplasm / nucleus類似検索 - 分子機能 Linker histone H1/H5 / linker histone H1 and H5 family / Linker histone H1/H5, domain H15 / Linker histone H1/H5 globular (H15) domain profile. / Domain in histone families 1 and 5 / Winged helix-like DNA-binding domain superfamily/Winged helix DNA-binding domain / Arc Repressor Mutant, subunit A / Winged helix DNA-binding domain superfamily / Winged helix-like DNA-binding domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Martinsen, J.H. / Bugge, K. / Kragelund, B.B. |
|---|
| 資金援助 |  デンマーク, 1件 デンマーク, 1件 | 組織 | 認可番号 | 国 |
|---|
| Novo Nordisk Foundation | |  デンマーク デンマーク |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Micromolar affinity association of an IDP and a folded protein without the involvement of persistent binding sites
著者: Bugge, K. / Martinsen, J.H. / Fernandes, C.B. / Borgia, A. / Schuler, B. / Best, R. / Kragelund, B.B. |
|---|
| 履歴 | | 登録 | 2018年9月23日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年10月9日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.2 | 2024年6月19日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 デンマーク, 1件
デンマーク, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6hq1.cif.gz
6hq1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6hq1.ent.gz
pdb6hq1.ent.gz PDB形式
PDB形式 6hq1.json.gz
6hq1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6hq1_validation.pdf.gz
6hq1_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6hq1_full_validation.pdf.gz
6hq1_full_validation.pdf.gz 6hq1_validation.xml.gz
6hq1_validation.xml.gz 6hq1_validation.cif.gz
6hq1_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/hq/6hq1
https://data.pdbj.org/pub/pdb/validation_reports/hq/6hq1 ftp://data.pdbj.org/pub/pdb/validation_reports/hq/6hq1
ftp://data.pdbj.org/pub/pdb/validation_reports/hq/6hq1 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: H1F0, H1FV / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: H1F0, H1FV / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj







 gel filtration
gel filtration