詳細 タイプ Solution-ID 内容 Label 溶媒系 solution1 1.2 mM unlabeled TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 95% H2O/5% D2O unlabeled_sample95% H2O/5% D2Osolution2 1.2 mM [U-13C; U-15N] TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 95% H2O/5% D2O 13C-15N_sample_H2O 95% H2O/5% D2Osolution3 1.2 mM [U-15N] TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 95% H2O/5% D2O 15N_sample95% H2O/5% D2Osolution4 1.2 mM [U-13C; U-15N]-Ade,Cyt TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 95% H2O/5% D2O A,C-13C-15N_sample_H2O 95% H2O/5% D2Osolution5 1.2 mM [U-13C; U-15N]-Gua,Ura TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 95% H2O/5% D2O G,U-13C-15N_sample_H2O 95% H2O/5% D2Osolution6 1.2 mM [U-13C; U-15N]-Ade,Cyt TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 100% D2O A,C-13C-15N_sample_D2O 100% D2Osolution7 1.2 mM [U-13C; U-15N]-Gua,Ura TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 100% D2O G,U-13C-15N_sample_D2O 100% D2Osolution8 1.2 mM [U-13C; U-15N]-Ade TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 95% H2O/5% D2O A-13C-15N-sample_H2O 95% H2O/5% D2Osolution9 1.2 mM [U-13C; U-15N] TMR3_48nt, 1.4 mM unlabeled 5-TAMRA, 100% D2O 13C-15N_sample_D2O 100% D2O
試料 大きな表を表示 (4 x 18) 大きな表を隠す 濃度 (mg/ml)構成要素 Isotopic labeling Solution-ID 1.2 mMTMR3_48ntunlabeled1 1.4 mM5-TAMRA unlabeled1 1.2 mMTMR3_48nt[U-13C; U-15N] 2 1.4 mM5-TAMRA unlabeled2 1.2 mMTMR3_48nt[U-15N] 3 1.4 mM5-TAMRA unlabeled3 1.2 mMTMR3_48nt[U-13C; U-15N]-Ade,Cyt 4 1.4 mM5-TAMRA unlabeled4 1.2 mMTMR3_48nt[U-13C; U-15N]-Gua,Ura 5 1.4 mM5-TAMRA unlabeled5 1.2 mMTMR3_48nt[U-13C; U-15N]-Ade,Cyt 6 1.4 mM5-TAMRA unlabeled6 1.2 mMTMR3_48nt[U-13C; U-15N]-Gua,Ura 7 1.4 mM5-TAMRA unlabeled7 1.2 mMTMR3_48nt[U-13C; U-15N]-Ade 8 1.4 mM5-TAMRA unlabeled8 1.2 mMTMR3_48nt[U-13C; U-15N] 9 1.4 mM5-TAMRA unlabeled9
試料状態 Conditions-ID イオン強度 Label pH 圧 (kPa)温度 (K)1 130 mM K-Glutamate, 15 mM mM Glutamate buffer 283K7.5 1 bar283 K2 130 mM K-Glutamate, 15 mM NaCl mM Glutamate buffer 298 K7.5 1 bar298 K
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 ドイツ, 1件
ドイツ, 1件  引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2020
ジャーナル: Nucleic Acids Res. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6gzr.cif.gz
6gzr.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6gzr.ent.gz
pdb6gzr.ent.gz PDB形式
PDB形式 6gzr.json.gz
6gzr.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6gzr_validation.pdf.gz
6gzr_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6gzr_full_validation.pdf.gz
6gzr_full_validation.pdf.gz 6gzr_validation.xml.gz
6gzr_validation.xml.gz 6gzr_validation.cif.gz
6gzr_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/gz/6gzr
https://data.pdbj.org/pub/pdb/validation_reports/gz/6gzr ftp://data.pdbj.org/pub/pdb/validation_reports/gz/6gzr
ftp://data.pdbj.org/pub/pdb/validation_reports/gz/6gzr リンク
リンク 集合体
集合体
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー



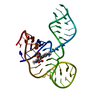

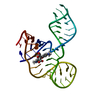
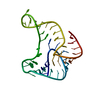


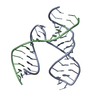



 PDBj
PDBj






























 CYANA
CYANA