+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5kh8 | ||||||
|---|---|---|---|---|---|---|---|
| Title | Solution structures of the apo state fluoride riboswitch | ||||||
 Components Components | riboswitch (47-MER) | ||||||
 Keywords Keywords | RNA / apo state / pseudoknot / riboswitch aptamer / transcriptional regulation | ||||||
| Function / homology | RNA / RNA (> 10) Function and homology information Function and homology information | ||||||
| Biological species | synthetic construct (others) | ||||||
| Method | SOLUTION NMR | ||||||
 Authors Authors | Zhang, Q. / Zhao, B. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: Nat. Chem. Biol. / Year: 2017 Journal: Nat. Chem. Biol. / Year: 2017Title: An excited state underlies gene regulation of a transcriptional riboswitch. Authors: Zhao, B. / Guffy, S.L. / Williams, B. / Zhang, Q. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5kh8.cif.gz 5kh8.cif.gz | 567.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5kh8.ent.gz pdb5kh8.ent.gz | 488.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5kh8.json.gz 5kh8.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/kh/5kh8 https://data.pdbj.org/pub/pdb/validation_reports/kh/5kh8 ftp://data.pdbj.org/pub/pdb/validation_reports/kh/5kh8 ftp://data.pdbj.org/pub/pdb/validation_reports/kh/5kh8 | HTTPS FTP |
|---|
-Related structure data
| Similar structure data | |
|---|---|
| Other databases |
- Links
Links
- Assembly
Assembly
| Deposited unit | 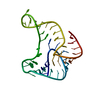
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: RNA chain | Mass: 15053.960 Da / Num. of mol.: 1 / Source method: obtained synthetically / Source: (synth.) synthetic construct (others) |
|---|
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR experiment |
|
- Sample preparation
Sample preparation
| Details |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Sample |
|
 Movie
Movie Controller
Controller











 PDBj
PDBj





























 HSQC
HSQC