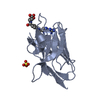
Entry Database : PDB / ID : 6gzpTitle Llama nanobody PorM_02 structure determined at room temperature by in-situ diffraction in ChipX microfluidic device Nanobody Keywords / / / Biological species Lama glama (llama)Method / / / Resolution : 2.1 Å Authors Roche, J. / Gaubert, A. / Desmyter, A. / De Wijn, R. / Sauter, C. / Roussel, A. Funding support Organization Grant number Country French National Research Agency
Journal : Iucrj / Year : 2019Title : A simple and versatile microfluidic device for efficient biomacromolecule crystallization and structural analysis by serial crystallography.Authors: de Wijn, R. / Hennig, O. / Roche, J. / Engilberge, S. / Rollet, K. / Fernandez-Millan, P. / Brillet, K. / Betat, H. / Morl, M. / Roussel, A. / Girard, E. / Mueller-Dieckmann, C. / Fox, G.C. ... Authors : de Wijn, R. / Hennig, O. / Roche, J. / Engilberge, S. / Rollet, K. / Fernandez-Millan, P. / Brillet, K. / Betat, H. / Morl, M. / Roussel, A. / Girard, E. / Mueller-Dieckmann, C. / Fox, G.C. / Olieric, V. / Gavira, J.A. / Lorber, B. / Sauter, C. History Deposition Jul 4, 2018 Deposition site / Processing site Revision 1.0 Jul 18, 2018 Provider / Type Revision 1.1 May 1, 2019 Group Data collection / Database references ... Data collection / Database references / Experimental preparation / Structure summary Category citation / database_PDB_rev ... citation / database_PDB_rev / database_PDB_rev_record / exptl_crystal_grow / pdbx_database_proc / struct Item _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.year / _exptl_crystal_grow.pdbx_details / _struct.title Revision 1.2 May 29, 2019 Group / Database references / Category / citation_author / pdbx_database_procItem _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_PubMed / _citation.title Revision 1.3 Jan 17, 2024 Group Advisory / Data collection ... Advisory / Data collection / Database references / Refinement description Category chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_unobs_or_zero_occ_atoms Item / _database_2.pdbx_database_accessionRevision 1.4 Nov 20, 2024 Group / Category / pdbx_modification_feature
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords
 X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.1 Å
MOLECULAR REPLACEMENT / Resolution: 2.1 Å  Authors
Authors France, 1items
France, 1items  Citation
Citation Journal: Iucrj / Year: 2019
Journal: Iucrj / Year: 2019 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 6gzp.cif.gz
6gzp.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb6gzp.ent.gz
pdb6gzp.ent.gz PDB format
PDB format 6gzp.json.gz
6gzp.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/gz/6gzp
https://data.pdbj.org/pub/pdb/validation_reports/gz/6gzp ftp://data.pdbj.org/pub/pdb/validation_reports/gz/6gzp
ftp://data.pdbj.org/pub/pdb/validation_reports/gz/6gzp




 Links
Links Assembly
Assembly
 Components
Components

 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  SOLEIL
SOLEIL  / Beamline: PROXIMA 2 / Wavelength: 0.826561272144 Å
/ Beamline: PROXIMA 2 / Wavelength: 0.826561272144 Å Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller











 PDBj
PDBj

