| 登録情報 | データベース: PDB / ID: 6f0a
|
|---|
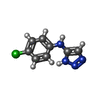
| タイトル | Crystal structure of human indoleamine 2,3-dioxygenase bound to a triazole inhibitor and alanine molecule. |
|---|
 要素 要素 | Indoleamine 2,3-dioxygenase 1 |
|---|
 キーワード キーワード | OXIDOREDUCTASE / Tryptophan 2 3 Dioxygenase Activity Electron Transfer Activity Oxidoreductase Activity Heme Binding Indoleamine 2 3 Dioxygenase Activity Metal Ion Binding Dioxygenase Activity |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
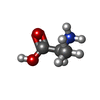
indoleamine 2,3-dioxygenase / positive regulation of chronic inflammatory response / smooth muscle contractile fiber / indoleamine 2,3-dioxygenase activity / kynurenic acid biosynthetic process / tryptophan 2,3-dioxygenase activity / positive regulation of T cell tolerance induction / L-tryptophan catabolic process to kynurenine / quinolinate biosynthetic process / stereocilium bundle ... indoleamine 2,3-dioxygenase / positive regulation of chronic inflammatory response / smooth muscle contractile fiber / indoleamine 2,3-dioxygenase activity / kynurenic acid biosynthetic process / tryptophan 2,3-dioxygenase activity / positive regulation of T cell tolerance induction / L-tryptophan catabolic process to kynurenine / quinolinate biosynthetic process / stereocilium bundle / 'de novo' NAD+ biosynthetic process from L-tryptophan / positive regulation of type 2 immune response / L-tryptophan catabolic process / negative regulation of T cell apoptotic process / Tryptophan catabolism / positive regulation of T cell apoptotic process / swimming behavior / negative regulation of interleukin-10 production / multicellular organismal response to stress / T cell proliferation / negative regulation of T cell proliferation / positive regulation of interleukin-12 production / female pregnancy / response to lipopolysaccharide / electron transfer activity / inflammatory response / heme binding / metal ion binding / cytosol / cytoplasm類似検索 - 分子機能 Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #480 / Indoleamine 2,3-dioxygenase / Indoleamine 2,3-dioxygenase / Indoleamine 2,3-dioxygenase signature 1. / Indoleamine 2,3-dioxygenase signature 2. / Tryptophan/Indoleamine 2,3-dioxygenase-like / Methane Monooxygenase Hydroxylase; Chain G, domain 1 / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 ALANINE / ~{N}-(4-chlorophenyl)-1~{H}-1,2,3-triazol-5-amine / PROTOPORPHYRIN IX CONTAINING FE / Indoleamine 2,3-dioxygenase 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.26 Å 分子置換 / 解像度: 2.26 Å |
|---|
 データ登録者 データ登録者 | Swan, M.K. / Latchem, M. |
|---|
 引用 引用 |  ジャーナル: Chembiochem / 年: 2018 ジャーナル: Chembiochem / 年: 2018
タイトル: New 4-Amino-1,2,3-Triazole Inhibitors of Indoleamine 2,3-Dioxygenase Form a Long-Lived Complex with the Enzyme and Display Exquisite Cellular Potency.
著者: Alexandre, J.A.C. / Swan, M.K. / Latchem, M.J. / Boyall, D. / Pollard, J.R. / Hughes, S.W. / Westcott, J. |
|---|
| 履歴 | | 登録 | 2017年11月17日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年12月27日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年2月7日 | Group: Database references / カテゴリ: citation / citation_author / Item: _citation.title / _citation_author.name |
|---|
| 改定 1.2 | 2018年4月4日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.year |
|---|
| 改定 1.3 | 2019年10月16日 | Group: Data collection / カテゴリ: reflns / reflns_shell / Item: _reflns.pdbx_CC_half / _reflns_shell.pdbx_CC_half |
|---|
| 改定 1.4 | 2024年1月17日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_label_asym_id |
|---|
| 改定 1.5 | 2024年11月6日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.26 Å
分子置換 / 解像度: 2.26 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chembiochem / 年: 2018
ジャーナル: Chembiochem / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6f0a.cif.gz
6f0a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6f0a.ent.gz
pdb6f0a.ent.gz PDB形式
PDB形式 6f0a.json.gz
6f0a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/f0/6f0a
https://data.pdbj.org/pub/pdb/validation_reports/f0/6f0a ftp://data.pdbj.org/pub/pdb/validation_reports/f0/6f0a
ftp://data.pdbj.org/pub/pdb/validation_reports/f0/6f0a
 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: IDO1, IDO, INDO / プラスミド: pET28 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: IDO1, IDO, INDO / プラスミド: pET28 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.9763 Å
/ ビームライン: I03 / 波長: 0.9763 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj