| 登録情報 | データベース: PDB / ID: 6cmn
|
|---|
| タイトル | Co-Crystal Structure of HIV-1 TAR Bound to Lab-Evolved RRM TBP6.7 |
|---|
 要素 要素 | - TAR-Binding Protein 6.7
- Trans-Activation Response RNA Element
|
|---|
 キーワード キーワード | RNA BINDING PROTEIN/RNA / protein-RNA complex / TAR RNA / lab-evolved protein / arginine fork / beta hairpin / major-groove readout / base triple / U1A / HIV-1 / trans-activation / RNA Recognition Motif / RRM / RNA BINDING PROTEIN-RNA complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
U1 snRNP / U1 snRNA binding / U4/U6 x U5 tri-snRNP complex / spliceosomal complex / mRNA splicing, via spliceosome / nucleoplasm / identical protein binding類似検索 - 分子機能 U1 small nuclear ribonucleoprotein A, RNA recognition motif 1 / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain / RNA-binding domain superfamily / Nucleotide-binding alpha-beta plait domain superfamily / Alpha-Beta Plaits / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 RNA / RNA (> 10) / Small nuclear ribonucleoprotein polypeptide A類似検索 - 構成要素 |
|---|
| 生物種 |   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ)
  Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.796 Å 分子置換 / 解像度: 1.796 Å |
|---|
 データ登録者 データ登録者 | Belashov, I.A. / Wedekind, J.E. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM123864 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2018 ジャーナル: Nucleic Acids Res. / 年: 2018
タイトル: Structure of HIV TAR in complex with a Lab-Evolved RRM provides insight into duplex RNA recognition and synthesis of a constrained peptide that impairs transcription.
著者: Belashov, I.A. / Crawford, D.W. / Cavender, C.E. / Dai, P. / Beardslee, P.C. / Mathews, D.H. / Pentelute, B.L. / McNaughton, B.R. / Wedekind, J.E. |
|---|
| 履歴 | | 登録 | 2018年3月5日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2018年6月6日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年7月11日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2018年8月8日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.3 | 2019年2月20日 | Group: Author supporting evidence / Data collection / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2020年1月1日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.5 | 2023年10月4日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.796 Å
分子置換 / 解像度: 1.796 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2018
ジャーナル: Nucleic Acids Res. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6cmn.cif.gz
6cmn.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6cmn.ent.gz
pdb6cmn.ent.gz PDB形式
PDB形式 6cmn.json.gz
6cmn.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6cmn_validation.pdf.gz
6cmn_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6cmn_full_validation.pdf.gz
6cmn_full_validation.pdf.gz 6cmn_validation.xml.gz
6cmn_validation.xml.gz 6cmn_validation.cif.gz
6cmn_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/cm/6cmn
https://data.pdbj.org/pub/pdb/validation_reports/cm/6cmn ftp://data.pdbj.org/pub/pdb/validation_reports/cm/6cmn
ftp://data.pdbj.org/pub/pdb/validation_reports/cm/6cmn
 リンク
リンク 集合体
集合体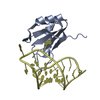
 要素
要素


 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRL
SSRL  / ビームライン: BL12-2 / 波長: 0.9795 Å
/ ビームライン: BL12-2 / 波長: 0.9795 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



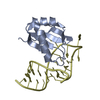









 PDBj
PDBj































