| 登録情報 | データベース: PDB / ID: 6c6m
|
|---|
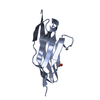
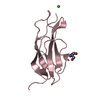
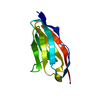
| タイトル | IgCam3 of human MLCK1 |
|---|
 要素 要素 | Myosin light chain kinase, smooth muscle |
|---|
 キーワード キーワード | CELL ADHESION / IgCAM MLCK1 IBD |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
aorta smooth muscle tissue morphogenesis / tonic smooth muscle contraction / myosin-light-chain kinase / myosin light chain kinase activity / cellular hypotonic response / bleb assembly / positive regulation of calcium ion transport / positive regulation of wound healing / RHO GTPases activate PAKs / cleavage furrow ...aorta smooth muscle tissue morphogenesis / tonic smooth muscle contraction / myosin-light-chain kinase / myosin light chain kinase activity / cellular hypotonic response / bleb assembly / positive regulation of calcium ion transport / positive regulation of wound healing / RHO GTPases activate PAKs / cleavage furrow / smooth muscle contraction / regulation of synaptic vesicle endocytosis / Smooth Muscle Contraction / stress fiber / actin cytoskeleton / lamellipodium / actin binding / scaffold protein binding / calmodulin binding / protein phosphorylation / positive regulation of cell migration / synapse / ATP binding / metal ion binding / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 Myosin Light Chain Kinase 1, Kinase domain / Unstructured linker between I-set domains 2 and 3 on MYLCK / Immunoglobulin I-set / Immunoglobulin I-set domain / Fibronectin type III domain / Fibronectin type 3 domain / Immunoglobulin subtype 2 / Immunoglobulin C-2 Type / Fibronectin type-III domain profile. / Fibronectin type III ...Myosin Light Chain Kinase 1, Kinase domain / Unstructured linker between I-set domains 2 and 3 on MYLCK / Immunoglobulin I-set / Immunoglobulin I-set domain / Fibronectin type III domain / Fibronectin type 3 domain / Immunoglobulin subtype 2 / Immunoglobulin C-2 Type / Fibronectin type-III domain profile. / Fibronectin type III / Fibronectin type III superfamily / Immunoglobulin subtype / Immunoglobulin / Serine/threonine-protein kinase, active site / Serine/Threonine protein kinases active-site signature. / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily / Protein kinase domain / Serine/Threonine protein kinases, catalytic domain / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Immunoglobulin-like fold / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.5 Å シンクロトロン / 解像度: 2.5 Å |
|---|
 データ登録者 データ登録者 | Zuccola, H.J. / Turner, J. |
|---|
| 資金援助 |  米国, 2件 米国, 2件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of Diabetes and Digestive and Kidney Disease (NIH/NIDDK) | R01DK61931 |  米国 米国 | | National Institutes of Health/National Institute of Diabetes and Digestive and Kidney Disease (NIH/NIDDK) | R01DK068271 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Nat. Med. / 年: 2019 ジャーナル: Nat. Med. / 年: 2019
タイトル: Intracellular MLCK1 diversion reverses barrier loss to restore mucosal homeostasis.
著者: Graham, W.V. / He, W. / Marchiando, A.M. / Zha, J. / Singh, G. / Li, H.S. / Biswas, A. / Ong, M.L.D.M. / Jiang, Z.H. / Choi, W. / Zuccola, H. / Wang, Y. / Griffith, J. / Wu, J. / Rosenberg, H. ...著者: Graham, W.V. / He, W. / Marchiando, A.M. / Zha, J. / Singh, G. / Li, H.S. / Biswas, A. / Ong, M.L.D.M. / Jiang, Z.H. / Choi, W. / Zuccola, H. / Wang, Y. / Griffith, J. / Wu, J. / Rosenberg, H.J. / Wang, Y. / Snapper, S.B. / Ostrov, D. / Meredith, S.C. / Miller, L.W. / Turner, J.R. |
|---|
| 履歴 | | 登録 | 2018年1月19日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2019年1月23日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年8月7日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.journal_abbrev / _citation.journal_id_CSD ..._citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2019年12月25日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.3 | 2024年3月6日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / software
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _software.name |
|---|
| 改定 1.4 | 2024年11月6日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.5 Å
シンクロトロン / 解像度: 2.5 Å  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Nat. Med. / 年: 2019
ジャーナル: Nat. Med. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6c6m.cif.gz
6c6m.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6c6m.ent.gz
pdb6c6m.ent.gz PDB形式
PDB形式 6c6m.json.gz
6c6m.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6c6m_validation.pdf.gz
6c6m_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6c6m_full_validation.pdf.gz
6c6m_full_validation.pdf.gz 6c6m_validation.xml.gz
6c6m_validation.xml.gz 6c6m_validation.cif.gz
6c6m_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/c6/6c6m
https://data.pdbj.org/pub/pdb/validation_reports/c6/6c6m ftp://data.pdbj.org/pub/pdb/validation_reports/c6/6c6m
ftp://data.pdbj.org/pub/pdb/validation_reports/c6/6c6m リンク
リンク 集合体
集合体



 要素
要素 Homo sapiens (ヒト) / 遺伝子: MYLK, MLCK, MLCK1, MYLK1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: MYLK, MLCK, MLCK1, MYLK1 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.2 / 波長: 1 Å
/ ビームライン: 5.0.2 / 波長: 1 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj








