登録情報 データベース : PDB / ID : 6b0lタイトル KLP10A-AMPPNP in complex with a microtubule Kinesin-like protein Klp10A Tubulin alpha-1B chain Tubulin beta chain キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Drosophila melanogaster (キイロショウジョウバエ)Sus scrofa (ブタ)手法 / / / 解像度 : 3.98 Å データ登録者 Benoit, M.P.M.H. / Asenjo, A.B. / Sosa, H. 資金援助 組織 認可番号 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) R01GM113164
ジャーナル : Nat Commun / 年 : 2018タイトル : Cryo-EM reveals the structural basis of microtubule depolymerization by kinesin-13s.著者 : Matthieu P M H Benoit / Ana B Asenjo / Hernando Sosa / 要旨 : Kinesin-13s constitute a distinct group within the kinesin superfamily of motor proteins that promote microtubule depolymerization and lack motile activity. The molecular mechanism by which kinesin- ... Kinesin-13s constitute a distinct group within the kinesin superfamily of motor proteins that promote microtubule depolymerization and lack motile activity. The molecular mechanism by which kinesin-13s depolymerize microtubules and are adapted to perform a seemingly very different activity from other kinesins is still unclear. To address this issue, here we report the near atomic resolution cryo-electron microscopy (cryo-EM) structures of Drosophila melanogaster kinesin-13 KLP10A protein constructs bound to curved or straight tubulin in different nucleotide states. These structures show how nucleotide induced conformational changes near the catalytic site are coupled with movement of the kinesin-13-specific loop-2 to induce tubulin curvature leading to microtubule depolymerization. The data highlight a modular structure that allows similar kinesin core motor-domains to be used for different functions, such as motility or microtubule depolymerization. 履歴 登録 2017年9月14日 登録サイト / 処理サイト 改定 1.0 2018年5月2日 Provider / タイプ 改定 1.1 2018年5月9日 Group / Database references / カテゴリ Item _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_PubMed / _citation.title 改定 1.2 2020年1月1日 Group / カテゴリ / Item 改定 1.3 2024年3月13日 Group / Database references / Derived calculationsカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_struct_oper_list Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_oper_list.name / _pdbx_struct_oper_list.symmetry_operation / _pdbx_struct_oper_list.type
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2018
ジャーナル: Nat Commun / 年: 2018
 構造の表示
構造の表示 ムービービューア
ムービービューア Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6b0l.cif.gz
6b0l.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6b0l.ent.gz
pdb6b0l.ent.gz PDB形式
PDB形式 6b0l.json.gz
6b0l.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/b0/6b0l
https://data.pdbj.org/pub/pdb/validation_reports/b0/6b0l ftp://data.pdbj.org/pub/pdb/validation_reports/b0/6b0l
ftp://data.pdbj.org/pub/pdb/validation_reports/b0/6b0l リンク
リンク 集合体
集合体


 要素
要素






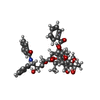



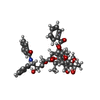

 試料調製
試料調製
 電子顕微鏡撮影
電子顕微鏡撮影
 FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM
FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





















