| 登録情報 | データベース: PDB / ID: 6ahy
|
|---|
| タイトル | Wnt signaling complex |
|---|
 要素 要素 | - Frizzled-8
- Proto-oncogene Wnt-3
|
|---|
 キーワード キーワード | SIGNALING PROTEIN / Wnt signaling / Canonical Wnt pathway |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
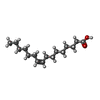
Spemann organizer formation at the anterior end of the primitive streak / positive regulation of collateral sprouting in absence of injury / regulation of mesenchymal stem cell differentiation / Wnt-Frizzled-LRP5/6 complex / Regulation of FZD by ubiquitination / Asymmetric localization of PCP proteins / WNT ligand biogenesis and trafficking / canonical Wnt signaling pathway involved in mesenchymal stem cell differentiation / negative regulation of axon extension involved in axon guidance / Wnt receptor activity ...Spemann organizer formation at the anterior end of the primitive streak / positive regulation of collateral sprouting in absence of injury / regulation of mesenchymal stem cell differentiation / Wnt-Frizzled-LRP5/6 complex / Regulation of FZD by ubiquitination / Asymmetric localization of PCP proteins / WNT ligand biogenesis and trafficking / canonical Wnt signaling pathway involved in mesenchymal stem cell differentiation / negative regulation of axon extension involved in axon guidance / Wnt receptor activity / limb bud formation / dorsal/ventral axis specification / gamete generation / Wnt-protein binding / mammary gland epithelium development / midbrain dopaminergic neuron differentiation / head morphogenesis / frizzled binding / embryonic forelimb morphogenesis / Class B/2 (Secretin family receptors) / Wnt signalosome / embryonic hindlimb morphogenesis / anterior/posterior axis specification / mesoderm formation / cell fate commitment / positive regulation of Wnt signaling pathway / regulation of neurogenesis / canonical Wnt signaling pathway / positive regulation of osteoblast differentiation / neuronal dense core vesicle / cellular response to retinoic acid / TCF dependent signaling in response to WNT / extracellular matrix / axon guidance / cytokine activity / stem cell proliferation / PDZ domain binding / G protein-coupled receptor activity / Golgi lumen / Wnt signaling pathway / endocytic vesicle membrane / neuron differentiation / cell morphogenesis / T cell differentiation in thymus / angiogenesis / gene expression / receptor ligand activity / endoplasmic reticulum lumen / protein domain specific binding / signaling receptor binding / ubiquitin protein ligase binding / positive regulation of gene expression / Golgi apparatus / extracellular space / extracellular exosome / extracellular region / plasma membrane類似検索 - 分子機能 Wnt-3 protein / Frizzled 8, cysteine-rich domain / Wnt protein, conserved site / Wnt-1 family signature. / Wnt / Wnt, C-terminal domain / wnt family / found in Wnt-1 / Frizzled cysteine-rich domain / Frizzled cysteine-rich domain ...Wnt-3 protein / Frizzled 8, cysteine-rich domain / Wnt protein, conserved site / Wnt-1 family signature. / Wnt / Wnt, C-terminal domain / wnt family / found in Wnt-1 / Frizzled cysteine-rich domain / Frizzled cysteine-rich domain / Frizzled/Smoothened, transmembrane domain / Frizzled/Smoothened family membrane region / Frizzled/Smoothened family membrane region / Frizzled/secreted frizzled-related protein / Frizzled / Frizzled domain / Frizzled cysteine-rich domain superfamily / Fz domain / Frizzled (fz) domain profile. / GPCR, family 2-like / G-protein coupled receptors family 2 profile 2. / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 PALMITOLEIC ACID / Proto-oncogene Wnt-3 / Frizzled-8類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ)
 Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å |
|---|
 データ登録者 データ登録者 | Hirai, H. / Arimori, T. / Matoba, K. / Mihara, E. / Takagi, J. |
|---|
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2019 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2019
タイトル: Crystal structure of a mammalian Wnt-frizzled complex.
著者: Hirai, H. / Matoba, K. / Mihara, E. / Arimori, T. / Takagi, J. |
|---|
| 履歴 | | 登録 | 2018年8月21日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年5月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年5月15日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.pdbx_database_id_PubMed / _citation.title ..._citation.pdbx_database_id_PubMed / _citation.title / _citation_author.identifier_ORCID / _citation_author.name |
|---|
| 改定 1.2 | 2019年5月22日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_unobs_or_zero_occ_atoms / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.occupancy / _atom_site.type_symbol / _chem_comp.name / _chem_comp.type / _pdbx_entity_nonpoly.entity_id / _pdbx_entity_nonpoly.name / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_unobs_or_zero_occ_atoms.label_asym_id / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2023年11月22日 | Group: Data collection / Database references ...Data collection / Database references / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.8 Å
分子置換 / 解像度: 2.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2019
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6ahy.cif.gz
6ahy.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6ahy.ent.gz
pdb6ahy.ent.gz PDB形式
PDB形式 6ahy.json.gz
6ahy.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6ahy_validation.pdf.gz
6ahy_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6ahy_full_validation.pdf.gz
6ahy_full_validation.pdf.gz 6ahy_validation.xml.gz
6ahy_validation.xml.gz 6ahy_validation.cif.gz
6ahy_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ah/6ahy
https://data.pdbj.org/pub/pdb/validation_reports/ah/6ahy ftp://data.pdbj.org/pub/pdb/validation_reports/ah/6ahy
ftp://data.pdbj.org/pub/pdb/validation_reports/ah/6ahy
 リンク
リンク 集合体
集合体



 要素
要素
 Homo sapiens (ヒト) / 参照: UniProt: Q61091
Homo sapiens (ヒト) / 参照: UniProt: Q61091 Homo sapiens (ヒト) / 遺伝子: WNT3, INT4 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: WNT3, INT4 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P56703
Homo sapiens (ヒト) / 参照: UniProt: P56703


 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL44XU / 波長: 0.9 Å
/ ビームライン: BL44XU / 波長: 0.9 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





