[English] 日本語
 Yorodumi
Yorodumi- PDB-5ywy: Crystal structure of the human prostaglandin E receptor EP4 in co... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5ywy | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of the human prostaglandin E receptor EP4 in complex with Fab and ONO-AE3-208 | |||||||||
 Components Components |
| |||||||||
 Keywords Keywords | SIGNALING PROTEIN-IMMUNE SYSTEM / G-protein coupled receptor / lipid mediator / functional antibody / SIGNALING PROTEIN-IMMUNE SYSTEM complex / MEMBRANE PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of eosinophil extravasation / prostaglandin E receptor activity / negative regulation of integrin activation / Prostanoid ligand receptors / T-helper cell differentiation / regulation of stress fiber assembly / negative regulation of cytokine production / response to mechanical stimulus / JNK cascade / ERK1 and ERK2 cascade ...negative regulation of eosinophil extravasation / prostaglandin E receptor activity / negative regulation of integrin activation / Prostanoid ligand receptors / T-helper cell differentiation / regulation of stress fiber assembly / negative regulation of cytokine production / response to mechanical stimulus / JNK cascade / ERK1 and ERK2 cascade / response to prostaglandin E / positive regulation of cytokine production / cellular response to mechanical stimulus / bone development / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / negative regulation of inflammatory response / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / positive regulation of inflammatory response / cellular response to prostaglandin E stimulus / positive regulation of cytosolic calcium ion concentration / G alpha (s) signalling events / immune response / inflammatory response / membrane / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 3.2 Å MOLECULAR REPLACEMENT / Resolution: 3.2 Å | |||||||||
 Authors Authors | Toyoda, Y. / Morimoto, K. / Suno, R. / Horita, S. / Iwata, S. / Kobayashi, T. | |||||||||
| Funding support |  Japan, 2items Japan, 2items
| |||||||||
 Citation Citation |  Journal: Nat. Chem. Biol. / Year: 2019 Journal: Nat. Chem. Biol. / Year: 2019Title: Ligand binding to human prostaglandin E receptor EP4at the lipid-bilayer interface. Authors: Toyoda, Y. / Morimoto, K. / Suno, R. / Horita, S. / Yamashita, K. / Hirata, K. / Sekiguchi, Y. / Yasuda, S. / Shiroishi, M. / Shimizu, T. / Urushibata, Y. / Kajiwara, Y. / Inazumi, T. / ...Authors: Toyoda, Y. / Morimoto, K. / Suno, R. / Horita, S. / Yamashita, K. / Hirata, K. / Sekiguchi, Y. / Yasuda, S. / Shiroishi, M. / Shimizu, T. / Urushibata, Y. / Kajiwara, Y. / Inazumi, T. / Hotta, Y. / Asada, H. / Nakane, T. / Shiimura, Y. / Nakagita, T. / Tsuge, K. / Yoshida, S. / Kuribara, T. / Hosoya, T. / Sugimoto, Y. / Nomura, N. / Sato, M. / Hirokawa, T. / Kinoshita, M. / Murata, T. / Takayama, K. / Yamamoto, M. / Narumiya, S. / Iwata, S. / Kobayashi, T. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5ywy.cif.gz 5ywy.cif.gz | 302.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5ywy.ent.gz pdb5ywy.ent.gz | 243.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5ywy.json.gz 5ywy.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/yw/5ywy https://data.pdbj.org/pub/pdb/validation_reports/yw/5ywy ftp://data.pdbj.org/pub/pdb/validation_reports/yw/5ywy ftp://data.pdbj.org/pub/pdb/validation_reports/yw/5ywy | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5yfiC  5yhlC C: citing same article ( |
|---|---|
| Similar structure data | |
| Experimental dataset #1 | Data reference:  10.5281/zenodo.1173791 / Data set type: diffraction image data / Details: Raw data 10.5281/zenodo.1173791 / Data set type: diffraction image data / Details: Raw data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 37063.547 Da / Num. of mol.: 1 Mutation: N7Q, A62L, G106R, N177Q, 218-259 deletion,N7Q, A62L, G106R, N177Q, 218-259 deletion Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PTGER4, PTGER2 / Production host: Homo sapiens (human) / Gene: PTGER4, PTGER2 / Production host:  |
|---|---|
| #2: Antibody | Mass: 27574.092 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  |
| #3: Antibody | Mass: 26614.857 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  |
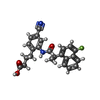
| #4: Chemical | ChemComp-7UR / |
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.29 Å3/Da / Density % sol: 71.34 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: lipidic cubic phase Details: 24-31% PEG 300, 150 mM potassium sulfate, 100 mM MES pH 5.5-6.5, 1% 1,2,3-heptantoriol PH range: 5.5-6.5 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SPring-8 SPring-8  / Beamline: BL32XU / Wavelength: 1 Å / Beamline: BL32XU / Wavelength: 1 Å |
| Detector | Type: RAYONIX MX225-HS / Detector: CCD / Date: Oct 3, 2015 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 3.2→50 Å / Num. obs: 26463 / % possible obs: 99.9 % / Redundancy: 52 % / CC1/2: 0.997 / Net I/σ(I): 9 |
| Reflection shell | Resolution: 3.2→3.33 Å |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 3.2→48.879 Å / SU ML: 0.39 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 24.13 MOLECULAR REPLACEMENT / Resolution: 3.2→48.879 Å / SU ML: 0.39 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 24.13
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 253.38 Å2 / Biso mean: 82.0147 Å2 / Biso min: 14.76 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 3.2→48.879 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 9
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj