| 登録情報 | データベース: PDB / ID: 5xof
|
|---|
| タイトル | Crystal structure of human paired immunoglobulin-like type 2 receptor alpha with synthesized glycopeptide I |
|---|
 要素 要素 | - Paired immunoglobulin-like type 2 receptor alpha
- Peptide from Nitric oxide synthase, endothelial
|
|---|
 キーワード キーワード | IMMUNE SYSTEM / membrane protein / immune receptor / viral entry inhibitor / glycopeptide |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of the force of heart contraction by chemical signal / NOSIP mediated eNOS trafficking / negative regulation of muscle hyperplasia / tetrahydrobiopterin metabolic process / NOSTRIN mediated eNOS trafficking / smooth muscle hyperplasia / regulation of nervous system process / superoxide-generating NAD(P)H oxidase activity / ovulation from ovarian follicle / pulmonary valve morphogenesis ...regulation of the force of heart contraction by chemical signal / NOSIP mediated eNOS trafficking / negative regulation of muscle hyperplasia / tetrahydrobiopterin metabolic process / NOSTRIN mediated eNOS trafficking / smooth muscle hyperplasia / regulation of nervous system process / superoxide-generating NAD(P)H oxidase activity / ovulation from ovarian follicle / pulmonary valve morphogenesis / response to fluid shear stress / negative regulation of biomineral tissue development / Nitric oxide stimulates guanylate cyclase / regulation of systemic arterial blood pressure by endothelin / ROS and RNS production in phagocytes / tetrahydrobiopterin binding / arginine binding / aortic valve morphogenesis / endocardial cushion morphogenesis / ventricular septum morphogenesis / positive regulation of Notch signaling pathway / cadmium ion binding / negative regulation of calcium ion transport / MHC class I protein binding / negative regulation of potassium ion transport / negative regulation of platelet activation / blood vessel remodeling / nitric oxide mediated signal transduction / actin monomer binding / nitric-oxide synthase (NADPH) / positive regulation of blood vessel endothelial cell migration / nitric-oxide synthase activity / endothelial cell migration / L-arginine catabolic process / negative regulation of extrinsic apoptotic signaling pathway via death domain receptors / regulation of sodium ion transport / eNOS activation / nitric oxide metabolic process / response to hormone / Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / negative regulation of blood pressure / nitric oxide biosynthetic process / homeostasis of number of cells within a tissue / removal of superoxide radicals / cell redox homeostasis / lipopolysaccharide-mediated signaling pathway / blood vessel diameter maintenance / VEGFR2 mediated vascular permeability / mitochondrion organization / negative regulation of smooth muscle cell proliferation / lung development / establishment of localization in cell / potassium ion transport / caveola / regulation of blood pressure / positive regulation of angiogenesis / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / calcium ion transport / endocytic vesicle membrane / vasodilation / FMN binding / flavin adenine dinucleotide binding / NADP binding / response to heat / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / scaffold protein binding / angiogenesis / response to lipopolysaccharide / in utero embryonic development / cytoskeleton / Extra-nuclear estrogen signaling / calmodulin binding / Golgi membrane / negative regulation of cell population proliferation / heme binding / positive regulation of gene expression / Golgi apparatus / signal transduction / extracellular exosome / nucleus / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 : / Nitric-oxide synthase, eukaryote / Nitric oxide synthase, N-terminal / Nitric oxide synthase, N-terminal domain superfamily / Nitric oxide synthase, domain 2 superfamily / Nitric oxide synthase, domain 1 superfamily / Nitric oxide synthase, domain 3 superfamily / : / Nitric oxide synthase, oxygenase domain / Nitric oxide synthase (NOS) signature. ...: / Nitric-oxide synthase, eukaryote / Nitric oxide synthase, N-terminal / Nitric oxide synthase, N-terminal domain superfamily / Nitric oxide synthase, domain 2 superfamily / Nitric oxide synthase, domain 1 superfamily / Nitric oxide synthase, domain 3 superfamily / : / Nitric oxide synthase, oxygenase domain / Nitric oxide synthase (NOS) signature. / Sulfite reductase [NADPH] flavoprotein alpha-component-like, FAD-binding / NADPH-cytochrome p450 reductase, FAD-binding, alpha-helical domain superfamily / FAD binding domain / Flavodoxin-like / Flavoprotein pyridine nucleotide cytochrome reductase / Flavodoxin / Flavodoxin-like domain profile. / Flavodoxin/nitric oxide synthase / Oxidoreductase FAD/NAD(P)-binding / Oxidoreductase NAD-binding domain / FAD-binding domain, ferredoxin reductase-type / Ferredoxin-NADP reductase (FNR), nucleotide-binding domain / Ferredoxin reductase-type FAD binding domain profile. / Riboflavin synthase-like beta-barrel / Flavoprotein-like superfamily / Immunoglobulin V-set domain / Immunoglobulin V-set domain / Immunoglobulin subtype / Immunoglobulin / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily / Immunoglobulin-like fold / Immunoglobulins / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 Nitric oxide synthase 3 / Paired immunoglobulin-like type 2 receptor alpha類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.963 Å 分子置換 / 解像度: 1.963 Å |
|---|
 データ登録者 データ登録者 | Furukawa, A. / Kakita, K. / Yamada, T. / Ishizuka, M. / Sakamoto, J. / Hatori, N. / Maeda, N. / Ohsaka, F. / Saitoh, T. / Nomura, T. ...Furukawa, A. / Kakita, K. / Yamada, T. / Ishizuka, M. / Sakamoto, J. / Hatori, N. / Maeda, N. / Ohsaka, F. / Saitoh, T. / Nomura, T. / Kuroki, K. / Nambu, H. / Arase, H. / Matsunaga, H. / Anada, M. / Ose, T. / Hashimoto, S. / Maenaka, K. |
|---|
 引用 引用 |  ジャーナル: J. Biol. Chem. / 年: 2017 ジャーナル: J. Biol. Chem. / 年: 2017
タイトル: Structural and thermodynamic analyses reveal critical features of glycopeptide recognition by the human PILR alpha immune cell receptor.
著者: Furukawa, A. / Kakita, K. / Yamada, T. / Ishizuka, M. / Sakamoto, J. / Hatori, N. / Maeda, N. / Ohsaka, F. / Saitoh, T. / Nomura, T. / Kuroki, K. / Nambu, H. / Arase, H. / Matsunaga, S. / ...著者: Furukawa, A. / Kakita, K. / Yamada, T. / Ishizuka, M. / Sakamoto, J. / Hatori, N. / Maeda, N. / Ohsaka, F. / Saitoh, T. / Nomura, T. / Kuroki, K. / Nambu, H. / Arase, H. / Matsunaga, S. / Anada, M. / Ose, T. / Hashimoto, S. / Maenaka, K. |
|---|
| 履歴 | | 登録 | 2017年5月28日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2017年10月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年1月24日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_validate_close_contact / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _chem_comp.name / _chem_comp.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_validate_close_contact.auth_asym_id_2 / _pdbx_validate_close_contact.auth_seq_id_2 / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2023年11月22日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 2.2 | 2024年11月13日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.963 Å
分子置換 / 解像度: 1.963 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J. Biol. Chem. / 年: 2017
ジャーナル: J. Biol. Chem. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5xof.cif.gz
5xof.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5xof.ent.gz
pdb5xof.ent.gz PDB形式
PDB形式 5xof.json.gz
5xof.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5xof_validation.pdf.gz
5xof_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5xof_full_validation.pdf.gz
5xof_full_validation.pdf.gz 5xof_validation.xml.gz
5xof_validation.xml.gz 5xof_validation.cif.gz
5xof_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xo/5xof
https://data.pdbj.org/pub/pdb/validation_reports/xo/5xof ftp://data.pdbj.org/pub/pdb/validation_reports/xo/5xof
ftp://data.pdbj.org/pub/pdb/validation_reports/xo/5xof リンク
リンク 集合体
集合体
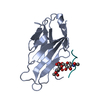

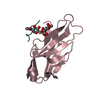

 要素
要素 Homo sapiens (ヒト) / 遺伝子: PILRA / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PILRA / 発現宿主: 
 Homo sapiens (ヒト) / 参照: UniProt: P29474, nitric-oxide synthase (NADPH)
Homo sapiens (ヒト) / 参照: UniProt: P29474, nitric-oxide synthase (NADPH) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL44XU / 波長: 1 Å
/ ビームライン: BL44XU / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー




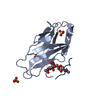










 PDBj
PDBj









