| 登録情報 | データベース: PDB / ID: 5vjj
|
|---|
| タイトル | Crystal structure of the flax-rust effector AvrP |
|---|
 要素 要素 | Avirulence protein AvrP123 |
|---|
 キーワード キーワード | METAL BINDING PROTEIN / flax rust (Melampsora lini) effector / nuclear localisation / plant disease resistance / zinc finger |
|---|
| 機能・相同性 | Avirulence protein AvrP123 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Melampsora lini (菌類) Melampsora lini (菌類) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.52 Å 多波長異常分散 / 解像度: 2.52 Å |
|---|
 データ登録者 データ登録者 | Zhang, X. / Ericsson, D.J. / Williams, S.J. / Kobe, B. |
|---|
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件 | 組織 | 認可番号 | 国 |
|---|
| Australian Research Council (ARC) | DP120100685, DP130104098 and DP160102244 |  オーストラリア オーストラリア |
|
|---|
 引用 引用 |  ジャーナル: Mol. Plant Pathol. / 年: 2018タイトル ジャーナル: Mol. Plant Pathol. / 年: 2018タイトル: Crystal structure of the Melampsora lini effector AvrP reveals insights into a possible nuclear function and recognition by the flax disease resistance protein P. 著者: Zhang, X. / Farah, N. / Rolston, L. / Ericsson, D.J. / Catanzariti, A.M. / Bernoux, M. / Ve, T. / Bendak, K. / Chen, C. / Mackay, J.P. / Lawrence, G.J. / Hardham, A. / Ellis, J.G. / Williams, ...著者: Zhang, X. / Farah, N. / Rolston, L. / Ericsson, D.J. / Catanzariti, A.M. / Bernoux, M. / Ve, T. / Bendak, K. / Chen, C. / Mackay, J.P. / Lawrence, G.J. / Hardham, A. / Ellis, J.G. / Williams, S.J. / Dodds, P.N. / Jones, D.A. / Kobe, B. |
|---|
| 履歴 | | 登録 | 2017年4月19日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年8月30日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年4月25日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.year |
|---|
| 改定 1.2 | 2020年1月1日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.3 | 2024年3月13日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Melampsora lini (菌類)
Melampsora lini (菌類) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 2.52 Å
多波長異常分散 / 解像度: 2.52 Å  データ登録者
データ登録者 オーストラリア, 1件
オーストラリア, 1件  引用
引用 ジャーナル: Mol. Plant Pathol. / 年: 2018
ジャーナル: Mol. Plant Pathol. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5vjj.cif.gz
5vjj.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5vjj.ent.gz
pdb5vjj.ent.gz PDB形式
PDB形式 5vjj.json.gz
5vjj.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5vjj_validation.pdf.gz
5vjj_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5vjj_full_validation.pdf.gz
5vjj_full_validation.pdf.gz 5vjj_validation.xml.gz
5vjj_validation.xml.gz 5vjj_validation.cif.gz
5vjj_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vj/5vjj
https://data.pdbj.org/pub/pdb/validation_reports/vj/5vjj ftp://data.pdbj.org/pub/pdb/validation_reports/vj/5vjj
ftp://data.pdbj.org/pub/pdb/validation_reports/vj/5vjj リンク
リンク 集合体
集合体

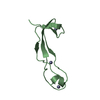
 要素
要素 Melampsora lini (菌類) / 発現宿主:
Melampsora lini (菌類) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX1 / 波長: 1.282, 1.262
/ ビームライン: MX1 / 波長: 1.282, 1.262 解析
解析 多波長異常分散 / 解像度: 2.52→37.01 Å / SU ML: 0.32 / 交差検証法: FREE R-VALUE / 位相誤差: 25.7
多波長異常分散 / 解像度: 2.52→37.01 Å / SU ML: 0.32 / 交差検証法: FREE R-VALUE / 位相誤差: 25.7  ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj


