[English] 日本語
 Yorodumi
Yorodumi- PDB-5uwl: Matrix metalloproteinase-13 complexed with selective inhibitor co... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5uwl | ||||||
|---|---|---|---|---|---|---|---|
| Title | Matrix metalloproteinase-13 complexed with selective inhibitor compound (S)-17a | ||||||
 Components Components | Collagenase 3 | ||||||
 Keywords Keywords | hydrolase/hydrolase inhibitor / Metalloproteinase / collagenase / MMP-13 / hydrolase / hydrolase-hydrolase inhibitor complex | ||||||
| Function / homology |  Function and homology information Function and homology informationgrowth plate cartilage development / RUNX2 regulates genes involved in cell migration / endochondral ossification / bone morphogenesis / Hydrolases; Acting on peptide bonds (peptidases); Metalloendopeptidases / Assembly of collagen fibrils and other multimeric structures / Activation of Matrix Metalloproteinases / bone mineralization / response to amyloid-beta / Collagen degradation ...growth plate cartilage development / RUNX2 regulates genes involved in cell migration / endochondral ossification / bone morphogenesis / Hydrolases; Acting on peptide bonds (peptidases); Metalloendopeptidases / Assembly of collagen fibrils and other multimeric structures / Activation of Matrix Metalloproteinases / bone mineralization / response to amyloid-beta / Collagen degradation / collagen catabolic process / extracellular matrix disassembly / collagen binding / Degradation of the extracellular matrix / extracellular matrix organization / metalloendopeptidase activity / extracellular matrix / endopeptidase activity / serine-type endopeptidase activity / calcium ion binding / proteolysis / extracellular space / extracellular region / zinc ion binding Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.55 Å MOLECULAR REPLACEMENT / Resolution: 2.55 Å | ||||||
 Authors Authors | Taylor, A.B. / Cao, X. / Hart, P.J. | ||||||
 Citation Citation |  Journal: J. Med. Chem. / Year: 2017 Journal: J. Med. Chem. / Year: 2017Title: Structure-Based Design and Synthesis of Potent and Selective Matrix Metalloproteinase 13 Inhibitors. Authors: Choi, J.Y. / Fuerst, R. / Knapinska, A.M. / Taylor, A.B. / Smith, L. / Cao, X. / Hart, P.J. / Fields, G.B. / Roush, W.R. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5uwl.cif.gz 5uwl.cif.gz | 83.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5uwl.ent.gz pdb5uwl.ent.gz | 61.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5uwl.json.gz 5uwl.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/uw/5uwl https://data.pdbj.org/pub/pdb/validation_reports/uw/5uwl ftp://data.pdbj.org/pub/pdb/validation_reports/uw/5uwl ftp://data.pdbj.org/pub/pdb/validation_reports/uw/5uwl | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5uwkC  5uwmC  5uwnC  4l19S C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 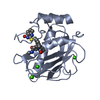
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 19366.578 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Details: CATALYTIC DOMAIN (UNP RESIDUES 104-274) / Source: (gene. exp.)  Homo sapiens (human) / Gene: MMP13 / Plasmid: PKA8H / Production host: Homo sapiens (human) / Gene: MMP13 / Plasmid: PKA8H / Production host:  References: UniProt: P45452, Hydrolases; Acting on peptide bonds (peptidases); Metalloendopeptidases #2: Chemical | #3: Chemical | ChemComp-CA / #4: Chemical | #5: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.47 Å3/Da / Density % sol: 72.49 % |
|---|---|
| Crystal grow | Temperature: 277 K / Method: vapor diffusion, sitting drop / pH: 7 Details: 0.2 M sodium chloride, 0.1 M Tris pH 7, 1.0 M sodium citrate |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 24-ID-E / Wavelength: 0.97918 Å / Beamline: 24-ID-E / Wavelength: 0.97918 Å |
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Nov 14, 2015 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.97918 Å / Relative weight: 1 |
| Reflection | Resolution: 2.55→60.07 Å / Num. obs: 23277 / % possible obs: 99 % / Redundancy: 7.3 % / Biso Wilson estimate: 50.1 Å2 / Rpim(I) all: 0.047 / Rsym value: 0.124 / Net I/σ(I): 12.1 |
| Reflection shell | Resolution: 2.55→2.69 Å / Redundancy: 7.4 % / Mean I/σ(I) obs: 2.1 / Num. unique obs: 3333 / Rpim(I) all: 0.369 / Rsym value: 0.967 / % possible all: 99.1 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4L19 Resolution: 2.55→60.07 Å / SU ML: 0.36 / Cross valid method: THROUGHOUT / σ(F): 1.35 / Phase error: 25.76
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.55→60.07 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller











 PDBj
PDBj











