+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5nir | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of collagen 2A vWC domain | ||||||
 要素 要素 | Collagen alpha-1(II) chain | ||||||
 キーワード キーワード | STRUCTURAL PROTEIN / collagen / vwc / ECM / BMP-2 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報collagen type II trimer / collagen type XI trimer / anterior head development / embryonic skeletal joint morphogenesis / otic vesicle development / Collagen chain trimerization / platelet-derived growth factor binding / extracellular matrix structural constituent conferring tensile strength / Extracellular matrix organization / notochord development ...collagen type II trimer / collagen type XI trimer / anterior head development / embryonic skeletal joint morphogenesis / otic vesicle development / Collagen chain trimerization / platelet-derived growth factor binding / extracellular matrix structural constituent conferring tensile strength / Extracellular matrix organization / notochord development / limb bud formation / proteoglycan metabolic process / cartilage development involved in endochondral bone morphogenesis / Collagen biosynthesis and modifying enzymes / endochondral ossification / MHC class II protein binding / Signaling by PDGF / cellular response to BMP stimulus / tissue homeostasis / NCAM1 interactions / collagen fibril organization / cartilage development / proteoglycan binding / Assembly of collagen fibrils and other multimeric structures / MET activates PTK2 signaling / inner ear morphogenesis / cartilage condensation / Collagen degradation / roof of mouth development / basement membrane / Non-integrin membrane-ECM interactions / chondrocyte differentiation / ECM proteoglycans / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / Integrin cell surface interactions / heart morphogenesis / extrinsic apoptotic signaling pathway in absence of ligand / visual perception / central nervous system development / skeletal system development / sensory perception of sound / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / : / regulation of gene expression / endoplasmic reticulum lumen / protein homodimerization activity / extracellular space / extracellular region / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.74 Å 単波長異常分散 / 解像度: 1.74 Å | ||||||
 データ登録者 データ登録者 | Fischer, G. / Blythe, E. / Hyvonen, M. | ||||||
 引用 引用 |  ジャーナル: J. Biol. Chem. / 年: 2017 ジャーナル: J. Biol. Chem. / 年: 2017タイトル: Structural analyses of von Willebrand factor C domains of collagen 2A and CCN3 reveal an alternative mode of binding to bone morphogenetic protein-2. 著者: Xu, E.R. / Blythe, E.E. / Fischer, G. / Hyvonen, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5nir.cif.gz 5nir.cif.gz | 47 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5nir.ent.gz pdb5nir.ent.gz | 32.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5nir.json.gz 5nir.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ni/5nir https://data.pdbj.org/pub/pdb/validation_reports/ni/5nir ftp://data.pdbj.org/pub/pdb/validation_reports/ni/5nir ftp://data.pdbj.org/pub/pdb/validation_reports/ni/5nir | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 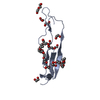
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 7921.925 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: COL2A1 / プラスミド: pHAT3 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: COL2A1 / プラスミド: pHAT3 / 発現宿主:  |
|---|
-非ポリマー , 5種, 38分子 








| #2: 化合物 | ChemComp-EDO / #3: 化合物 | ChemComp-PEG / #4: 化合物 | ChemComp-PG4 / #5: 化合物 | ChemComp-P33 / | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.83 Å3/Da / 溶媒含有率: 56.49 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: 0.1 M HEPES, pH 7.5, 40% PEG 200 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I24 / 波長: 0.96861 Å / ビームライン: I24 / 波長: 0.96861 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2013年1月30日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.96861 Å / 相対比: 1 |
| 反射 | 解像度: 1.74→49.37 Å / Num. obs: 17667 / % possible obs: 100 % / 冗長度: 6.4 % / Biso Wilson estimate: 30.54 Å2 / Rmerge(I) obs: 0.042 / Rsym value: 0.042 / Net I/σ(I): 22.7 |
| 反射 シェル | 解像度: 1.744→1.75 Å / 冗長度: 5.9 % / Rmerge(I) obs: 0.709 / Mean I/σ(I) obs: 2.2 / Rsym value: 0.709 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  単波長異常分散 / 解像度: 1.74→49.37 Å / Cor.coef. Fo:Fc: 0.947 / Cor.coef. Fo:Fc free: 0.937 / SU R Cruickshank DPI: 0.113 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.115 / SU Rfree Blow DPI: 0.107 / SU Rfree Cruickshank DPI: 0.106 単波長異常分散 / 解像度: 1.74→49.37 Å / Cor.coef. Fo:Fc: 0.947 / Cor.coef. Fo:Fc free: 0.937 / SU R Cruickshank DPI: 0.113 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.115 / SU Rfree Blow DPI: 0.107 / SU Rfree Cruickshank DPI: 0.106
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 39.26 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.26 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.74→49.37 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.74→1.84 Å / Rfactor Rfree error: 0 / Total num. of bins used: 9
|
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj
















