[English] 日本語
 Yorodumi
Yorodumi- PDB-5m5k: S-adenosyl-L-homocysteine hydrolase from Bradyrhizobium elkanii i... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5m5k | ||||||
|---|---|---|---|---|---|---|---|
| Title | S-adenosyl-L-homocysteine hydrolase from Bradyrhizobium elkanii in complex with adenosine and cordycepin | ||||||
 Components Components | Adenosylhomocysteinase | ||||||
 Keywords Keywords | HYDROLASE / SAHase / SAH / SAM-dependent methylation / nitrogen fixation / symbiotic bacteria / NAD / conformational transition / molecular gate / adenosine / cordycepin | ||||||
| Function / homology |  Function and homology information Function and homology informationL-homocysteine biosynthetic process / adenosylhomocysteinase / adenosylhomocysteinase activity / S-adenosylmethionine cycle / one-carbon metabolic process / cytosol Similarity search - Function | ||||||
| Biological species |  Bradyrhizobium elkanii (bacteria) Bradyrhizobium elkanii (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.84 Å MOLECULAR REPLACEMENT / Resolution: 1.84 Å | ||||||
 Authors Authors | Manszewski, T. / Mueller-Dieckamann, J. / Jaskolski, M. | ||||||
| Funding support |  Poland, 1items Poland, 1items
| ||||||
 Citation Citation |  Journal: IUCrJ / Year: 2017 Journal: IUCrJ / Year: 2017Title: Crystallographic and SAXS studies of S-adenosyl-l-homocysteine hydrolase from Bradyrhizobium elkanii. Authors: Manszewski, T. / Szpotkowski, K. / Jaskolski, M. #1:  Journal: Acta Crystallogr. D Biol. Crystallogr. / Year: 2015 Journal: Acta Crystallogr. D Biol. Crystallogr. / Year: 2015Title: An enzyme captured in two conformational states: crystal structure of S-adenosyl-L-homocysteine hydrolase from Bradyrhizobium elkanii. Authors: Manszewski, T. / Singh, K. / Imiolczyk, B. / Jaskolski, M. #2:  Journal: Acta Crystallogr. D Biol. Crystallogr. / Year: 2012 Journal: Acta Crystallogr. D Biol. Crystallogr. / Year: 2012Title: High-resolution structures of complexes of plant S-adenosyl-L-homocysteine hydrolase (Lupinus luteus). Authors: Brzezinski, K. / Dauter, Z. / Jaskolski, M. #3: Journal: Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun. Year: 2008 Title: Purification, crystallization and preliminary crystallographic studies of plant S-adenosyl-L-homocysteine hydrolase (Lupinus luteus). Authors: Brzezinski, K. / Bujacz, G. / Jaskolski, M. #4:  Journal: Nat. Struct. Biol. / Year: 1998 Journal: Nat. Struct. Biol. / Year: 1998Title: Structure determination of selenomethionyl S-adenosylhomocysteine hydrolase using data at a single wavelength. Authors: Turner, M.A. / Yuan, C.S. / Borchardt, R.T. / Hershfield, M.S. / Smith, G.D. / Howell, P.L. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5m5k.cif.gz 5m5k.cif.gz | 764.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5m5k.ent.gz pdb5m5k.ent.gz | 631 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5m5k.json.gz 5m5k.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/m5/5m5k https://data.pdbj.org/pub/pdb/validation_reports/m5/5m5k ftp://data.pdbj.org/pub/pdb/validation_reports/m5/5m5k ftp://data.pdbj.org/pub/pdb/validation_reports/m5/5m5k | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5m65C  5m66C  5m67C  4lvcS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | |
| Experimental dataset #1 | Data reference:  10.18150/repod.1236363 / Data set type: diffraction image data 10.18150/repod.1236363 / Data set type: diffraction image data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 4 molecules ABCD
| #1: Protein | Mass: 52737.238 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Bradyrhizobium elkanii (bacteria) / Gene: ahcY, BeSAHase / Production host: Bradyrhizobium elkanii (bacteria) / Gene: ahcY, BeSAHase / Production host:  |
|---|
-Non-polymers , 8 types, 1505 molecules 
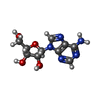


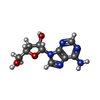










| #2: Chemical | ChemComp-NAD / #3: Chemical | #4: Chemical | #5: Chemical | #6: Chemical | ChemComp-3AD / | #7: Chemical | ChemComp-PGE / | #8: Chemical | ChemComp-ACT / | #9: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.32 Å3/Da / Density % sol: 46.87 % |
|---|---|
| Crystal grow | Temperature: 292 K / Method: vapor diffusion, sitting drop / pH: 8.5 Details: 0.2 M sodium acetate, 16% PEG 4000, 0.1 M Tris pH 8.5 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / Beamline: P14 (MX2) / Wavelength: 1.223 Å / Beamline: P14 (MX2) / Wavelength: 1.223 Å |
| Detector | Type: MAR555 FLAT PANEL / Detector: IMAGE PLATE / Date: May 31, 2012 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.223 Å / Relative weight: 1 |
| Reflection | Resolution: 1.84→47.82 Å / Num. obs: 168719 / % possible obs: 98.6 % / Redundancy: 7.6 % / Biso Wilson estimate: 21.26 Å2 / Rmerge(I) obs: 0.098 / Net I/σ(I): 16.62 |
| Reflection shell | Resolution: 1.84→1.95 Å / Redundancy: 5.08 % / Rmerge(I) obs: 1 / Mean I/σ(I) obs: 2.01 / % possible all: 91.7 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4LVC Resolution: 1.84→47.82 Å / SU ML: 0.15 / Cross valid method: FREE R-VALUE / Phase error: 17.96 Details: Riding H atoms were added. TLS parameters were included.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.84→47.82 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller


















 PDBj
PDBj








