Entry Database : PDB / ID : 5lvlTitle Human PDK1 Kinase Domain in Complex with Compound PS653 Bound to the ATP-Binding Site 3-phosphoinositide-dependent protein kinase 1 Keywords / / / / Function / homology Function Domain/homology Component
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / Biological species Homo sapiens (human)Method / / / Resolution : 1.4 Å Authors Schulze, J.O. / Saladino, G. / Busschots, K. / Neimanis, S. / Suess, E. / Odadzic, D. / Zeuzem, S. / Hindie, V. / Herbrand, A.K. / Lisa, M.N. ...Schulze, J.O. / Saladino, G. / Busschots, K. / Neimanis, S. / Suess, E. / Odadzic, D. / Zeuzem, S. / Hindie, V. / Herbrand, A.K. / Lisa, M.N. / Alzari, P.M. / Gervasio, F.L. / Biondi, R.M. Funding support Organization Grant number Country BMBF GO-Bio 0315102 German Research Foundation BI 1044/2-3 German Research Foundation BI 1044/12-1
Journal : Cell Chem Biol / Year : 2016Title : Bidirectional Allosteric Communication between the ATP-Binding Site and the Regulatory PIF Pocket in PDK1 Protein Kinase.Authors : Schulze, J.O. / Saladino, G. / Busschots, K. / Neimanis, S. / Su, E. / Odadzic, D. / Zeuzem, S. / Hindie, V. / Herbrand, A.K. / Lisa, M.N. / Alzari, P.M. / Gervasio, F.L. / Biondi, R.M. History Deposition Sep 14, 2016 Deposition site / Processing site Revision 1.0 Oct 19, 2016 Provider / Type Revision 1.1 Nov 2, 2016 Group Revision 2.0 Oct 16, 2019 Group / Author supporting evidence / Data collectionCategory / pdbx_audit_support / reflns_shellItem / _pdbx_audit_support.funding_organizationRevision 2.1 Jan 17, 2024 Group / Database references / Refinement descriptionCategory chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_database_accessionRevision 2.2 Nov 13, 2024 Group / Category / pdbx_modification_feature
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information Homo sapiens (human)
Homo sapiens (human) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.4 Å
MOLECULAR REPLACEMENT / Resolution: 1.4 Å  Authors
Authors Germany, 3items
Germany, 3items  Citation
Citation Journal: Cell Chem Biol / Year: 2016
Journal: Cell Chem Biol / Year: 2016 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 5lvl.cif.gz
5lvl.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb5lvl.ent.gz
pdb5lvl.ent.gz PDB format
PDB format 5lvl.json.gz
5lvl.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/lv/5lvl
https://data.pdbj.org/pub/pdb/validation_reports/lv/5lvl ftp://data.pdbj.org/pub/pdb/validation_reports/lv/5lvl
ftp://data.pdbj.org/pub/pdb/validation_reports/lv/5lvl




 Links
Links Assembly
Assembly
 Components
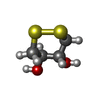
Components Homo sapiens (human) / Gene: PDPK1, PDK1 / Production host:
Homo sapiens (human) / Gene: PDPK1, PDK1 / Production host: 
 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  BESSY
BESSY  / Beamline: 14.1 / Wavelength: 0.91841 Å
/ Beamline: 14.1 / Wavelength: 0.91841 Å Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller












 PDBj
PDBj