| 登録情報 | データベース: PDB / ID: 5l4t
|
|---|
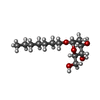
| タイトル | Crystal structure of FimH lectin domain in complex with 2-Deoxy-Heptylmannoside |
|---|
 要素 要素 | Protein FimH |
|---|
 キーワード キーワード | SUGAR BINDING PROTEIN / FimH / Type 1 pilus / urinary tract infection / UTI / carbohydrate / lectin / mannose / cell adhesion |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
pilus tip / mechanosensory behavior / cell adhesion involved in single-species biofilm formation / pilus / cell-substrate adhesion / D-mannose binding / host cell membrane / cell adhesion類似検索 - 分子機能 FimH, mannose-binding domain / FimH, mannose binding / Fimbrial-type adhesion domain / Fimbrial-type adhesion domain / Fimbrial protein / : / Fimbrial-type adhesion domain superfamily / Adhesion domain superfamily / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 heptyl 2-deoxy-alpha-D-mannopyranoside / Type 1 fimbrin D-mannose specific adhesin類似検索 - 構成要素 |
|---|
| 生物種 |   Escherichia coli K12 (大腸菌) Escherichia coli K12 (大腸菌)
  Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Jakob, R.P. / Zihlmann, P. / Rabbani, S. / Maier, T. / Ernst, B. |
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: High-Affinity Carbohydrate-Lectin Interactions: How Nature Makes it Possible
著者: Zihlmann, P. / Jiang, X. / Sager, C.P. / Fiege, B. / Jakob, R.P. / Siegrist, S. / Zalewski, A. / Rabbani, S. / Eris, D. / Silbermann, M. / Pang, L. / Muhlethaler, T. / Sharpe, T. / Maier, T. / Ernst, B. |
|---|
| 履歴 | | 登録 | 2016年5月26日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年6月21日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_entity_nonpoly / struct_site / struct_site_gen
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _atom_site.occupancy / _chem_comp.name / _chem_comp.pdbx_synonyms / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2020年8月5日 | Group: Derived calculations / Structure summary / カテゴリ: chem_comp / entity / pdbx_entity_nonpoly
Item: _chem_comp.name / _chem_comp.pdbx_synonyms ..._chem_comp.name / _chem_comp.pdbx_synonyms / _entity.pdbx_description / _pdbx_entity_nonpoly.name |
|---|
| 改定 2.2 | 2024年1月10日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 2.3 | 2024年11月6日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5l4t.cif.gz
5l4t.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5l4t.ent.gz
pdb5l4t.ent.gz PDB形式
PDB形式 5l4t.json.gz
5l4t.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5l4t_validation.pdf.gz
5l4t_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5l4t_full_validation.pdf.gz
5l4t_full_validation.pdf.gz 5l4t_validation.xml.gz
5l4t_validation.xml.gz 5l4t_validation.cif.gz
5l4t_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/l4/5l4t
https://data.pdbj.org/pub/pdb/validation_reports/l4/5l4t ftp://data.pdbj.org/pub/pdb/validation_reports/l4/5l4t
ftp://data.pdbj.org/pub/pdb/validation_reports/l4/5l4t リンク
リンク 集合体
集合体
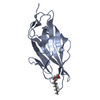

 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 1.00001 Å
/ ビームライン: X06SA / 波長: 1.00001 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー






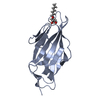
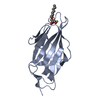


















 PDBj
PDBj