| 登録情報 | データベース: PDB / ID: 5kjy
|
|---|
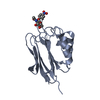
| タイトル | Co-crystal structure of PKA RI alpha CNB-B mutant (G316R/A336T) with cAMP |
|---|
 要素 要素 | cAMP-dependent protein kinase type I-alpha regulatory subunit |
|---|
 キーワード キーワード | SIGNALING PROTEIN / Cyclic nucleotide / cAMP-dependent protein kinase / nucleotide selectivity / cyclic nucleotide binding domain |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
sperm head-tail coupling apparatus / cAMP-dependent protein kinase regulator activity / nucleotide-activated protein kinase complex / ALK mutants bind TKIs / negative regulation of cAMP/PKA signal transduction / cAMP-dependent protein kinase inhibitor activity / sarcomere organization / cAMP-dependent protein kinase complex / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation ...sperm head-tail coupling apparatus / cAMP-dependent protein kinase regulator activity / nucleotide-activated protein kinase complex / ALK mutants bind TKIs / negative regulation of cAMP/PKA signal transduction / cAMP-dependent protein kinase inhibitor activity / sarcomere organization / cAMP-dependent protein kinase complex / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / ciliary base / negative regulation of activated T cell proliferation / protein kinase A catalytic subunit binding / mesoderm formation / plasma membrane raft / PKA activation in glucagon signalling / DARPP-32 events / cardiac muscle cell proliferation / cAMP binding / Hedgehog 'off' state / multivesicular body / cellular response to glucagon stimulus / FCGR3A-mediated IL10 synthesis / neuromuscular junction / positive regulation of insulin secretion / adenylate cyclase-activating G protein-coupled receptor signaling pathway / Signaling by ALK fusions and activated point mutants / Vasopressin regulates renal water homeostasis via Aquaporins / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / ADORA2B mediated anti-inflammatory cytokines production / GPER1 signaling / Factors involved in megakaryocyte development and platelet production / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / intracellular signal transduction / protein domain specific binding / negative regulation of gene expression / ubiquitin protein ligase binding / centrosome / regulation of transcription by RNA polymerase II / glutamatergic synapse / protein-containing complex / membrane / cytosol / cytoplasm類似検索 - 分子機能 cAMP-dependent protein kinase regulatory subunit / cAMP-dependent protein kinase regulatory subunit, dimerization-anchoring domain / Regulatory subunit of type II PKA R-subunit / RIIalpha, Regulatory subunit portion of type II PKA R-subunit / : / Cyclic nucleotide-binding domain signature 2. / Cyclic nucleotide-binding domain signature 1. / Cyclic nucleotide-binding, conserved site / Cyclic nucleotide-monophosphate binding domain / Cyclic nucleotide-binding domain ...cAMP-dependent protein kinase regulatory subunit / cAMP-dependent protein kinase regulatory subunit, dimerization-anchoring domain / Regulatory subunit of type II PKA R-subunit / RIIalpha, Regulatory subunit portion of type II PKA R-subunit / : / Cyclic nucleotide-binding domain signature 2. / Cyclic nucleotide-binding domain signature 1. / Cyclic nucleotide-binding, conserved site / Cyclic nucleotide-monophosphate binding domain / Cyclic nucleotide-binding domain / cAMP/cGMP binding motif profile. / Cyclic nucleotide-binding domain / Cyclic nucleotide-binding domain superfamily / Jelly Rolls / RmlC-like jelly roll fold / Jelly Rolls / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE / cAMP-dependent protein kinase type I-alpha regulatory subunit類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å |
|---|
 データ登録者 データ登録者 | Lorenz, R. / Moon, E. / Kim, J.J. / Huang, G.Y. / Kim, C. / Herberg, F.W. |
|---|
| 資金援助 |  ドイツ, ドイツ,  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Federal Ministry of Education and Research Grant | FKZ 0316177F (No Pain) |  ドイツ ドイツ | | The Center for Interdisciplinary Nanostructure Science and Technology (CINSaT) of the University of Kassel | |  ドイツ ドイツ | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | 2R01GM090161-06 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Biochem. J. / 年: 2017 ジャーナル: Biochem. J. / 年: 2017
タイトル: Mutations of PKA cyclic nucleotide-binding domains reveal novel aspects of cyclic nucleotide selectivity.
著者: Lorenz, R. / Moon, E.W. / Kim, J.J. / Schmidt, S.H. / Sankaran, B. / Pavlidis, I.V. / Kim, C. / Herberg, F.W. |
|---|
| 履歴 | | 登録 | 2016年6月20日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年6月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年7月19日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation_author.name |
|---|
| 改定 1.2 | 2017年8月2日 | Group: Structure summary / カテゴリ: struct / Item: _struct.title |
|---|
| 改定 1.3 | 2017年9月20日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2017年11月1日 | Group: Author supporting evidence / カテゴリ: pdbx_struct_assembly_auth_evidence |
|---|
| 改定 1.5 | 2019年12月25日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.6 | 2023年9月27日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 ドイツ,
ドイツ,  米国, 3件
米国, 3件  引用
引用 ジャーナル: Biochem. J. / 年: 2017
ジャーナル: Biochem. J. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5kjy.cif.gz
5kjy.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5kjy.ent.gz
pdb5kjy.ent.gz PDB形式
PDB形式 5kjy.json.gz
5kjy.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5kjy_validation.pdf.gz
5kjy_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5kjy_full_validation.pdf.gz
5kjy_full_validation.pdf.gz 5kjy_validation.xml.gz
5kjy_validation.xml.gz 5kjy_validation.cif.gz
5kjy_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/kj/5kjy
https://data.pdbj.org/pub/pdb/validation_reports/kj/5kjy ftp://data.pdbj.org/pub/pdb/validation_reports/kj/5kjy
ftp://data.pdbj.org/pub/pdb/validation_reports/kj/5kjy リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: PRKAR1A, PKR1, PRKAR1, TSE1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PRKAR1A, PKR1, PRKAR1, TSE1 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 31-ID / 波長: 0.97931 Å
/ ビームライン: 31-ID / 波長: 0.97931 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj