| 登録情報 | データベース: PDB / ID: 5ete
|
|---|
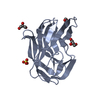
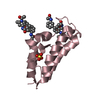
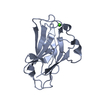
| タイトル | Structure of pathogen-related yeast protein, Pry1 in complex with a competitive inhibitor of cholesterol binding |
|---|
 要素 要素 | Pry1p |
|---|
 キーワード キーワード | Sterol binding protein / TAPs / testis specific proteins / Tpx /  antigen 5 (抗原) / Ag5 / antigen 5 (抗原) / Ag5 /  pathogenesis related-1 (感染特異的タンパク質) / PR-1 / Sc7 / CAP / pathogenesis related-1 (感染特異的タンパク質) / PR-1 / Sc7 / CAP /  cysteine-rich secretory protein / CRISP cysteine-rich secretory protein / CRISP |
|---|
| 機能・相同性 |  Pathogenesis-related Protein p14a / CAP / 3-Layer(aba) Sandwich / Alpha Beta / Pathogenesis-related Protein p14a / CAP / 3-Layer(aba) Sandwich / Alpha Beta /  1,4-ジオキサン / 1,4-ジオキサン /  : :  機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |    Saccharomyces cerevisiae YJM1434 (パン酵母) Saccharomyces cerevisiae YJM1434 (パン酵母) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å |
|---|
 データ登録者 データ登録者 | Asojo, O.A. |
|---|
 引用 引用 |  ジャーナル: Sci Rep / 年: 2016 ジャーナル: Sci Rep / 年: 2016
タイトル: Structural and functional characterization of the CAP domain of pathogen-related yeast 1 (Pry1) protein.
著者: Darwiche, R. / Kelleher, A. / Hudspeth, E.M. / Schneiter, R. / Asojo, O.A. |
|---|
| 履歴 | | 登録 | 2015年11月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年7月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年9月27日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_oper_list / pdbx_struct_special_symmetry
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_oper_list.symmetry_operation |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード antigen 5 (抗原) / Ag5 /
antigen 5 (抗原) / Ag5 /  pathogenesis related-1 (感染特異的タンパク質) / PR-1 / Sc7 / CAP /
pathogenesis related-1 (感染特異的タンパク質) / PR-1 / Sc7 / CAP /  cysteine-rich secretory protein / CRISP
cysteine-rich secretory protein / CRISP Pathogenesis-related Protein p14a / CAP / 3-Layer(aba) Sandwich / Alpha Beta /
Pathogenesis-related Protein p14a / CAP / 3-Layer(aba) Sandwich / Alpha Beta /  1,4-ジオキサン /
1,4-ジオキサン /  :
:  機能・相同性情報
機能・相同性情報
 Saccharomyces cerevisiae YJM1434 (パン酵母)
Saccharomyces cerevisiae YJM1434 (パン酵母) X線回折 /
X線回折 /  分子置換 / 解像度: 2.1 Å
分子置換 / 解像度: 2.1 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Sci Rep / 年: 2016
ジャーナル: Sci Rep / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ete.cif.gz
5ete.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ete.ent.gz
pdb5ete.ent.gz PDB形式
PDB形式 5ete.json.gz
5ete.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/et/5ete
https://data.pdbj.org/pub/pdb/validation_reports/et/5ete ftp://data.pdbj.org/pub/pdb/validation_reports/et/5ete
ftp://data.pdbj.org/pub/pdb/validation_reports/et/5ete リンク
リンク 集合体
集合体
 要素
要素
 Saccharomyces cerevisiae YJM1434 (パン酵母)
Saccharomyces cerevisiae YJM1434 (パン酵母)
 Pichia (菌類) / 参照: UniProt: A0A0C6AG41
Pichia (菌類) / 参照: UniProt: A0A0C6AG41 1,4-ジオキサン
1,4-ジオキサン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 回転陽極 / タイプ: RIGAKU FR-E+ SUPERBRIGHT / 波長: 1.54056 Å
回転陽極 / タイプ: RIGAKU FR-E+ SUPERBRIGHT / 波長: 1.54056 Å : 1.54056 Å / 相対比: 1
: 1.54056 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj


